Especificidad de detección
En la mayoría de los casos, el propósito del diseño de cebadores es maximizar la especificidad de la PCR.Esto viene determinado por la influencia más o menos predecible de muchas variables.Una variable importante es la secuencia en el extremo 3' del cebador.
Es importante destacar que es más probable que los ensayos de PCR diseñados para la especificidad mantengan una alta eficiencia en un amplio rango dinámico, porque el ensayo no produce productos de amplificación no específicos, compitiendo así con los reactivos de PCR o inhibiendo la reacción de amplificación principal.
Por supuesto, en algunos casos, la especificidad no es lo más importante, por ejemplo, cuando el objetivo es cuantificar patógenos estrechamente relacionados pero diferentes, se requieren estándares especiales de diseño, optimización y verificación.
La curva de fusión es un método estándar para evaluar la especificidad de los amplicones, al menos en términos de amplificar un solo objetivo.Sin embargo, debe enfatizarse que las curvas de fusión pueden ser engañosas porque, por ejemplo, pueden verse afectadas por los efectos combinados de cebadores subóptimos y bajas concentraciones de plantilla.
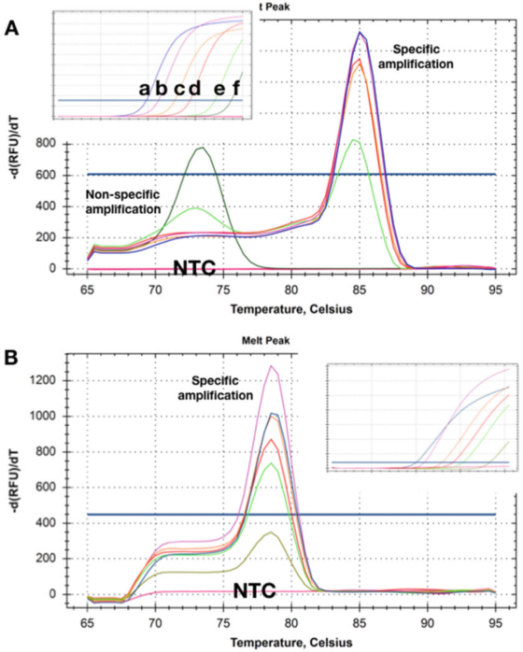
P5 |La curva de fusión muestra los cambios de Tm obtenidos a partir de dos detecciones de diferentes cantidades de dos ADN diana.
A. A concentraciones más altas (ad)), no hay un dímero de cebador obvio después de completar la medición de qPCR.A medida que la concentración de la plantilla disminuye a 50 copias (e), comienza a aparecer un producto no específico y se convierte en el único producto con la concentración más baja (f).
B. La prueba registró la misma Tms en todas las concentraciones objetivo y no hubo un dímero de cebador evidente incluso en la concentración más baja (5 copias).Al utilizar estos dos métodos de detección, no se detectaron productos de amplificación en los NTC.
P5 muestra las curvas de disolución obtenidas con muestras en las que el molde está presente a diferentes concentraciones.P 5a muestra que a las dos concentraciones más bajas, las Tms de los productos de amplificación no específicos producidos son más bajas que las de los amplicones específicos.
Obviamente, este método de detección no se puede utilizar de forma fiable para detectar objetivos que existen en bajas concentraciones.
Curiosamente, los NTC, es decir, muestras sin ADN en absoluto, no registraron productos de amplificación (no específicos), lo que indica que el ADN genómico de fondo puede participar en la amplificación/polimerización no específica.
A veces, estos cebadores de fondo y la amplificación no específica no se pueden remediar, pero a menudo es posible diseñar un método de detección que no tenga amplificación no específica en ninguna concentración de plantilla y NTC (P 5b).
Aquí, incluso registrar la amplificación de la concentración objetivo con un Cq de 35 producirá una curva de disolución específica.De manera similar, los NTC no mostraron signos de amplificación no específica.A veces, el comportamiento de detección puede depender de las aguas madres y solo se detecta una amplificación no específica en ciertas composiciones de tampón, lo que puede estar relacionado con diferentes concentraciones de Mg2+.
Estabilidad de detección
La optimización de Ta es un paso útil en el proceso de optimización y verificación empírica de la detección de qPCR.Proporciona una indicación directa de la solidez del conjunto de cebadores al mostrar la temperatura (o el rango de temperatura) que produce el Cq más bajo sin amplificar el NTC.
La diferencia de dos a cuatro veces en la sensibilidad puede no ser importante para las personas con alta expresión de ARNm, pero para las pruebas de diagnóstico, puede significar la diferencia entre resultados positivos y falsos negativos.
Las propiedades Ta de los cebadores de qPCR pueden variar mucho.Algunas pruebas no son muy sólidas y, si no se realizan con el valor Ta óptimo de los cebadores, colapsarán rápidamente.
Esto es importante porque este tipo de detección suele ser problemático en el mundo real, y la pureza de la muestra, la concentración de ADN o la presencia de otro ADN pueden no ser óptimas.
Además, el número de copias objetivo puede variar en un amplio rango, y los reactivos, utensilios de plástico o instrumentos pueden ser diferentes de los que se usaron al configurar la prueba.
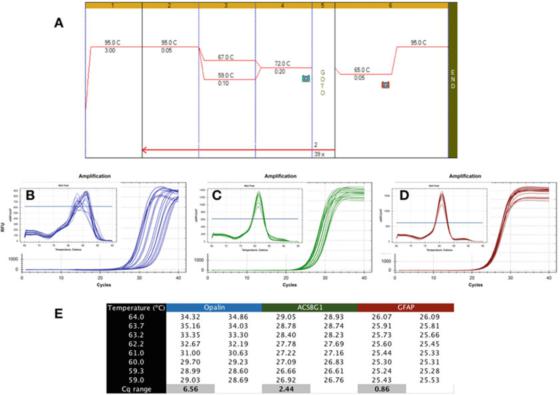
P6|El gradiente de temperatura muestra la diferente robustez de la detección por PCR.
A. Use la mezcla maestra Sensifast SYBR de Bioline (número de catálogo BIO-98050) para realizar PCR en ADNc preparado a partir de ARN de cerebro humano.
B. Utilice el instrumento CFX qPCR de Bio-Rad para registrar el mapa de amplificación y la curva de disolución de apaleno (NM_033207, F: GCCATGGAGGAAAGTGACAGACC, R: CTCATGTGTGGGTGATCTCCTAGG).
C. Gráfico de amplificación y curva de fusión de ACSBG1 (NM_015162.4, F: CTACACTTCCGGCACCACTGG, R: GTCCACGTGATATTGTCTTGACTCAG).
D. Gráfico de amplificación y curva de disolución de GFAP (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, R: CGAACCTCCTCCTCGTGGATCTTC).
E. Cqs registrado a diferentes temperaturas de recocido, mostrando la diferencia en Cq registrado bajo un gradiente de temperatura de 7C.
P 6 muestra un resultado típico de una prueba no deseada, en la que se realizó qPCR usando un gradiente Tas entre 59C y 67C (P 6a), usando cebadores para tres genes específicos del cerebro humano.
Se puede ver en el gráfico de amplificación que los cebadores Opalin están lejos de ser ideales porque su rango Ta óptimo es muy estrecho (Figura 6b), es decir, los Cq están muy dispersos, lo que hace que los Cq se comparen significativamente con su Cq bajo óptimo.
Este método de detección es inestable y puede conducir a una amplificación subóptima.Por lo tanto, este par de cebadores debería rediseñarse.Además, el análisis de la curva de fusión (recuadro) muestra que la especificidad de este método de detección también puede ser problemática, porque la curva de fusión de cada Ta es diferente.
El método de detección de ACSBG1 que se muestra en P 6c es más sólido que el método de detección de Opalin anterior, pero todavía está lejos de ser ideal y es probable que se pueda mejorar.
Sin embargo, enfatizamos que no existe una conexión necesaria entre robustez y especificidad, porque la curva de disolución producida por este método de detección muestra el mismo valor máximo en todos los Tas (recuadro).
Por otro lado, la prueba de robustez es mucho más tolerante, produciendo Cqs similares en una amplia gama de Tas, como en la prueba GFAP que se muestra en P 6d.
La diferencia en Cqs obtenida en el mismo rango de 8 grados Celsius es menor que 1, y la curva de disolución (recuadro) confirma las características de detección en este rango de temperatura.Vale la pena señalar que el Tas calculado y el rango Ta real pueden ser muy diferentes.
Hay muchas pautas diseñadas para ayudar a los investigadores a diseñar cebadores eficientes, la mayoría de las cuales se basan en reglas establecidas desde hace mucho tiempo y se ha prestado mucha atención al extremo 3' de los cebadores.A menudo se recomienda incluir una G o C en el extremo 3' y dos bases G o C (abrazadera GC), pero no más de dos de las últimas 5 bases.
En la práctica, estas reglas pueden guiar a los investigadores, pero no necesariamente son correctas en todas las circunstancias.
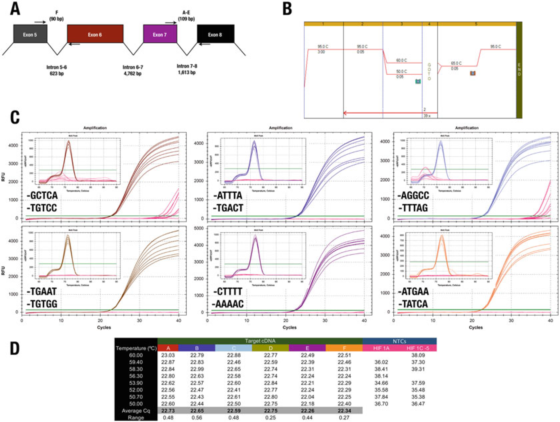
P7 |El extremo 3' del cebador tiene poco efecto sobre la especificidad o la eficiencia.
A. La posición de los cebadores para el gen humano HIF-1α (NM_181054.2).
B. Utilice licor madre Agilent Brilliant III SYBR Green (n.º de cat. 600882) para amplificar seis elementos de prueba.
C. Gráfico de amplificación y curva de fusión registrados por el instrumento CFX qPCR de Bio-Rad y los cebadores del extremo 3′.Los NTC se muestran en rojo.
D. Registro Cqs de cada elemento de prueba
Por ejemplo, el resultado en P 7 contradice la regla del extremo 3′.Todos los diseños producen básicamente los mismos resultados, con solo dos combinaciones de cebadores que conducen a una amplificación no específica en NTC.
Sin embargo, no podemos admitir el efecto del clip GC, porque en este caso, usar A o T como un máximo de 30 bases no reduce la especificidad.
La prueba C, donde el cebador F termina en GGCC, registró Cqs en NTC, lo que indica que es posible que desee evitar estas secuencias en el extremo 30.Hacemos hincapié en que la única forma de determinar la mejor secuencia del extremo 3' de un par de cebadores es evaluar experimentalmente algunos cebadores candidatos.
Eficiencia de amplificación
Es importante destacar que, aunque la detección de PCR no específica nunca puede volverse específica, la eficiencia de la amplificación se puede ajustar y maximizar de muchas maneras diferentes cambiando la enzima, las aguas madres, los aditivos y las condiciones del ciclo.
Para evaluar la eficacia de la detección por PCR, lo mejor es utilizar una dilución seriada de 10 o 5 veces el ácido nucleico objetivo, es decir, el “método de la curva estándar”.
Si se utilizan amplicones de PCR o dianas de ADN sintético para generar una curva estándar, las diluciones en serie de estas dianas deben mezclarse con una cantidad constante de ADN de fondo (como el ADN genómico).
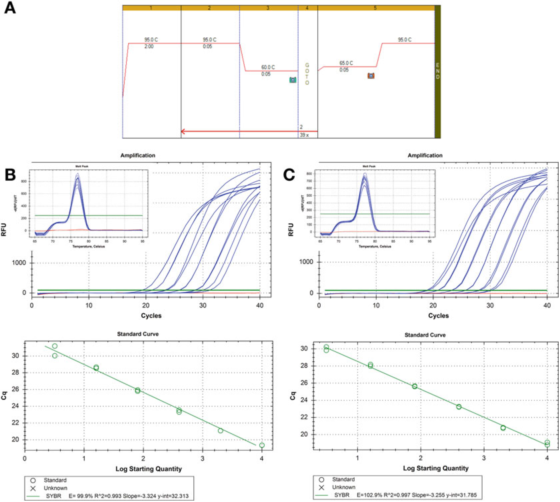
P8 |Curva de dilución para evaluar la eficiencia de la PCR.
A. Utilice cebadores para HIF-1: F: AAGAACTTTTAGGCCGTCCA y R: TTGTCCTGTGGTGACTTGTCC y la mezcla maestra Brilliant III SYBR Green de Agilent (número de catálogo 600882) para PCR y condiciones de curva de fusión.
B. 100 ng de ARN se transcribieron inversamente, se diluyeron 2 veces y las muestras de ADNc diluidas en serie se diluyeron 5 veces a 1 ng de ADN genómico humano.La curva de fusión se muestra en el recuadro.
C. La reacción de RT, la dilución y la dilución en serie se repitieron para la segunda muestra de ADNc y los resultados fueron similares.
P 8 muestra dos curvas estándar, utilizando el mismo método de detección en dos muestras de ADNc diferentes, el resultado es la misma eficiencia, alrededor del 100 %, y el valor de R2 también es similar, es decir, el grado de ajuste entre los datos experimentales y la línea de regresión o el grado de linealidad de los datos.
Las dos curvas estándar son comparables, pero no exactamente iguales.Si el propósito es cuantificar con precisión el objetivo, debe tenerse en cuenta que es inaceptable proporcionar un cálculo del número de copias sin explicar la incertidumbre.
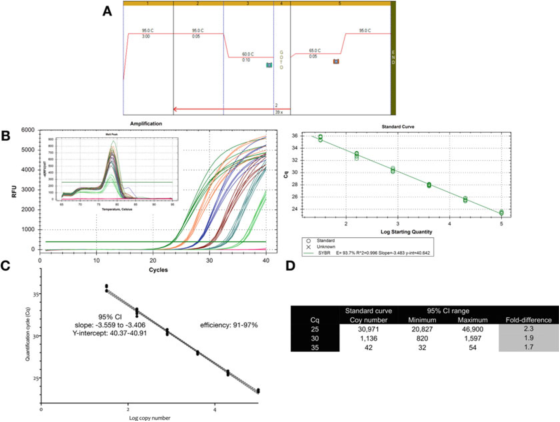
P9 |Incertidumbre de medida asociada a la cuantificación mediante una curva estándar.
A. Use cebadores para GAPDH (NM_002046) para realizar PCR y condiciones de curva de fusión.F: ACAGTTGCCATGTAGACC y R: TAACTGGTTGAGCACAGG y mezcla maestra Sensifast SYBR de Bioline (número de catálogo BIO-98050).
B. Gráfico de amplificación, curva de fusión y curva estándar registradas con el instrumento CFX qPCR de Bio-Rad.
C. Gráfico de curva estándar e intervalo de confianza (IC) del 95 %.
D. El número de copias y el intervalo de confianza del 95 % de los tres valores de Cq derivados de la curva de dilución.
P 9 muestra que para una prueba optimizada, la variabilidad inherente de una sola curva estándar es aproximadamente 2 veces (intervalo de confianza del 95%, mínimo a máximo), que puede ser la variabilidad más pequeña que se puede esperar.
Producto relacionado, artículo relacionado:
Kit qPCR de RT directa de células
Kit PCR directo de cola de ratón
Kit de PCR directa de tejido animal
Hora de publicación: 30-sep-2021