Las tres letras SNP son omnipresentes en el estudio de la genética de poblaciones.Independientemente de la investigación de enfermedades humanas, el posicionamiento de rasgos de cultivos, la evolución animal y la ecología molecular, los SNP son necesarios como base.Sin embargo, si no tiene un conocimiento profundo de la genética moderna basada en la secuenciación de alto rendimiento, y frente a estas tres letras, parece "el extraño más familiar", entonces no puede llevar a cabo la investigación posterior.Entonces, antes de embarcarnos en una investigación de seguimiento, echemos un vistazo a lo que es SNP.
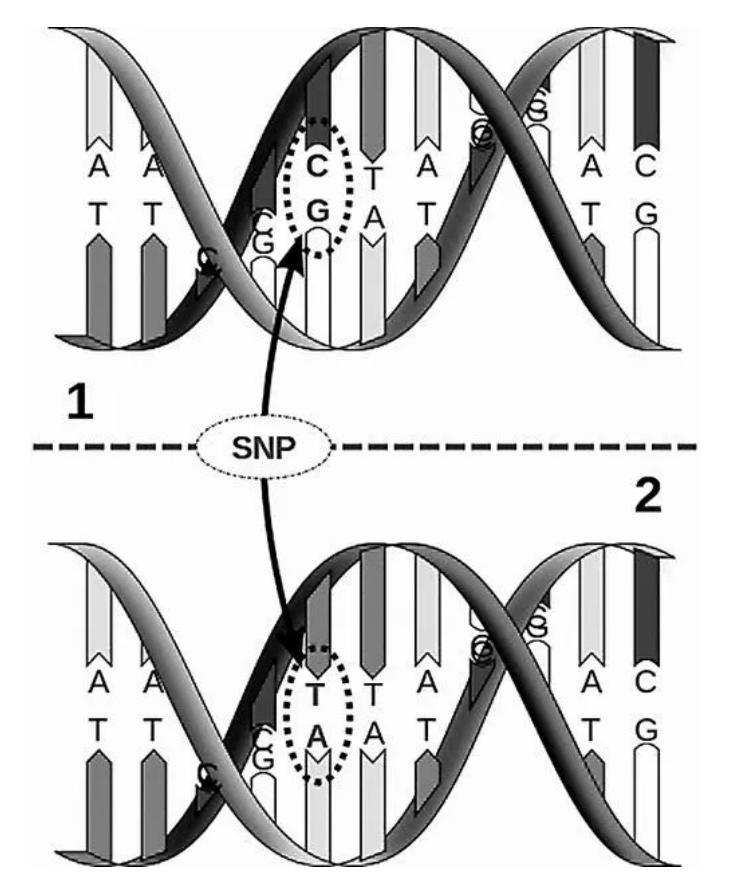
SNP (polimorfismo de un solo nucleótido), podemos ver por su nombre completo en inglés, se refiere a la variación o polimorfismo de un solo nucleótido.También tiene un nombre separado, llamado SNV (variación de un solo nucleótido).En algunos estudios en humanos, solo aquellos con una frecuencia poblacional superior al 1% se denominan SNP, pero en términos generales, los dos pueden mezclarse.Entonces podemos decir que SNP, polimorfismo de un solo nucleótido, se refiere a la mutación en la que un nucleótido en el genoma es reemplazado por otro nucleótido.Por ejemplo, en la siguiente figura, un par de bases AT se reemplaza con un par de bases GC, que es un sitio SNP.
Imagen
Sin embargo, ya sea un "polimorfismo de un solo nucleótido" o una "variación de un solo nucleótido", es relativamente hablando, por lo que los datos de SNP necesitan la resecuenciación del genoma como base, es decir, los datos de secuenciación se secuencian después de secuenciar el genoma del individuo.En comparación con el genoma, el sitio que difiere del genoma se detecta como un sitio SNP.
En términos de SNP en vegetales,el kit Plant Direct PCRse puede utilizar para la detección rápida.
En términos de tipos de mutación, SNP incluye transición y transversión.La transición se refiere a la sustitución de purinas por purinas o de pirimidinas por pirimidinas.La transversión se refiere a la sustitución mutua entre purinas y pirimidinas.La frecuencia de ocurrencia será diferente y la probabilidad de transición será mayor que la de transversión.
En términos de dónde ocurre el SNP, diferentes SNP tendrán diferentes efectos en el genoma.Los SNP que ocurren en la región intergénica, es decir, la región entre genes en el genoma, pueden no afectar la función del genoma, y las mutaciones en el intrón o en la región promotora aguas arriba del gen pueden tener cierto impacto en el gen;Las mutaciones que se producen en las regiones exónicas de los genes, según provoquen cambios en los aminoácidos codificados, tienen efectos diferentes sobre las funciones de los genes.(Por supuesto, incluso si dos SNP causan diferencias en los aminoácidos, tienen efectos diferentes en la estructura de la proteína y, en última instancia, los efectos en el fenotipo biológico pueden ser bastante diferentes).
Sin embargo, la cantidad de SNP que ocurren en ubicaciones de genes suele ser significativamente menor que la de ubicaciones sin genes, porque un SNP que afecta la función de un gen generalmente tiene un impacto negativo en la supervivencia del individuo, lo que resulta en que el individuo que porta este SNP en el grupo entre ellos sea eliminado.
Por supuesto, para los organismos diploides, los cromosomas existen en pares, pero es imposible que un par de cromosomas sea exactamente el mismo para cada base.Por lo tanto, algunos SNP también aparecerán heterocigotos, es decir, hay dos bases en esta posición en el cromosoma.En un grupo, los genotipos SNP de diferentes individuos se agregan, lo que se convierte en la base para la mayoría de los análisis posteriores.En combinación con los rasgos, se puede juzgar si SNP como marcador molecular está vinculado a los rasgos, se puede juzgar el QTL (locus de rasgos cuantitativos) del rasgo y se puede realizar GWAS (estudio de asociación de todo el genoma) o construcción de mapas genéticos;SNP se puede utilizar como marcador molecular Juzgar la relación evolutiva entre individuos;puede detectar SNP funcionales y estudiar mutaciones relacionadas con enfermedades;puede usar cambios de frecuencia de alelos SNP o tasas de heterocigosis y otros indicadores para determinar las regiones seleccionadas en el genoma... etc., combinado con el presente Con el desarrollo de la secuenciación de alto rendimiento, se pueden obtener cientos de miles o más sitios SNP a partir de un conjunto de datos de secuenciación.Se puede decir que SNP se ha convertido ahora en la piedra angular de la investigación genética de poblaciones.
Por supuesto, los cambios en las bases del genoma no siempre son el reemplazo de una base por otra (aunque esta es la más común).También es posible que falten una o algunas bases, o dos bases.Varias otras bases fueron insertadas en el medio.Esta pequeña gama de inserciones y eliminaciones se denomina colectivamente InDel (inserción y eliminación), que se refiere específicamente a la inserción y eliminación de fragmentos cortos (una o varias bases).InDel que ocurre en la ubicación de un gen también puede tener un impacto en la función del gen, por lo que a veces InDel también puede desempeñar un papel importante en la investigación.Pero, en general, el estatus de SNP como piedra angular de la genética de poblaciones sigue siendo inquebrantable.
Hora de publicación: 27 de agosto de 2021