1. Conocimientos básicos (si desea ver la parte experimental, transfiera directamente a la segunda parte)
Como una reacción derivada de la PCR convencional, la PCR en tiempo real monitorea principalmente el cambio de la cantidad de producto de amplificación en cada ciclo de la reacción de amplificación de PCR en tiempo real a través del cambio de la señal de fluorescencia y analiza cuantitativamente la plantilla inicial a través de la relación entre el valor ct y la curva estándar.
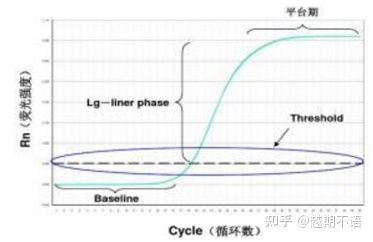
Los datos específicos de RT-PCR sonbase, umbral de fluorescenciayvalor CT.
| base: | El valor de fluorescencia del ciclo 3 al 15 es la línea de base (baseline), que es causada por el error ocasional de la medición. |
| Umbral (umbral): | Se refiere al límite de detección de fluorescencia establecido en una posición adecuada en la región de crecimiento exponencial de la curva de amplificación, generalmente 10 veces la desviación estándar de la línea de base. |
| Valor CT: | Es el número de ciclos de PCR cuando el valor de fluorescencia en cada tubo de reacción alcanza el umbral. El valor de Ct es inversamente proporcional a la cantidad de plantilla inicial. |
Métodos de etiquetado comunes para RT-PCR:
| método | ventaja | defecto | Ámbito de aplicación |
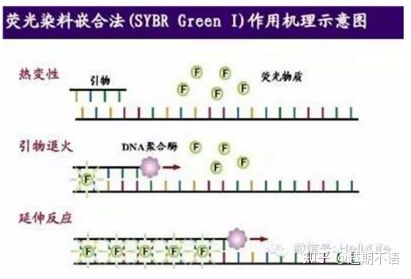
| SYBR VerdeⅠ | Amplia aplicabilidad, sensible, barato y conveniente. | Los requisitos de imprimación son altos, propensos a bandas no específicas | Es adecuado para el análisis cuantitativo de varios genes diana, la investigación sobre la expresión génica y la investigación sobre plantas y animales transgénicos recombinantes. |
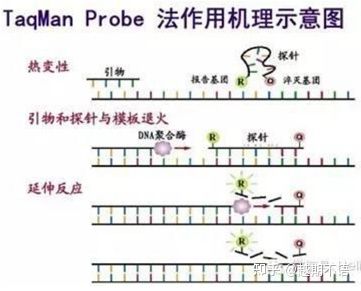
| TaqMan | Buena especificidad y alta repetibilidad. | El precio es alto y solo es adecuado para objetivos específicos. | Detección de patógenos, investigación de genes de resistencia a fármacos, evaluación de eficacia de fármacos, diagnóstico de enfermedades genéticas. |
| baliza molecular | Alta especificidad, fluorescencia, fondo bajo | El precio es alto, solo es adecuado para un propósito específico, el diseño es difícil y el precio es alto. | Análisis de genes específicos, análisis de SNP |
2. Pasos experimentales
2.1 Sobre el agrupamiento experimental- debe haber múltiples pozos en el grupo, y debe haber repeticiones biológicas.
| ① | Control en blanco | Se utiliza para detectar el estado de crecimiento celular en experimentos |
| ② | SiRNA de control negativo (secuencia de siRNA no específica) | Demostrar la especificidad de la acción del ARNi.El siRNA puede inducir una respuesta de estrés no específica a una concentración de 200 nM. |
| ③ | Control de reactivos de transfección | Excluir la toxicidad del reactivo de transfección para las células o el efecto sobre la expresión del gen diana |
| ④ | siRNA contra el gen diana | Derribar la expresión del gen objetivo |
| ⑤ (opcional) | ARNip positivo | Se utiliza para solucionar problemas operativos y del sistema experimental |
| ⑥ (opcional) | ARNip de control fluorescente | La eficiencia de la transfección celular se puede observar con un microscopio. |
2.2 Principios del diseño de cebadores
| Tamaño del fragmento amplificado | Preferiblemente a 100-150 pb |
| Longitud de imprimación | 18-25 pb |
| contenido de GC | 30%-70%, preferiblemente 45%-55% |
| valor tm | 58-60 ℃ |
| Secuencia | Evite T/C continuo;A/G continuo |
| 3 secuencia final | Evite los ricos en GC o AT;la base terminal es preferiblemente G o C;lo mejor es evitar T |
| complementariedad | Evite secuencias complementarias de más de 3 bases dentro del cebador o entre dos cebadores |
| especificidad | Utilice la búsqueda explosiva para confirmar la especificidad del cebador |
①SiRNA es específico de especie, y las secuencias de diferentes especies serán diferentes.
②SiRNA se envasa en polvo liofilizado, que se puede almacenar de forma estable durante 2-4 semanas a temperatura ambiente.
2.3 Herramientas o reactivos que deben prepararse con antelación
| Primer (referencia interna) | Incluyendo adelante y atrás dos |
| Cebadores (gen diana) | Incluyendo adelante y atrás dos |
| ARN de Si diana (3 tiras) | Generalmente, la empresa sintetizará 3 tiras y luego elegirá una de las tres por RT-PCR |
| Kit de transfección | Lipo2000 etc... |
| Kit de extracción rápida de ARN | Para la extracción de ARN después de la transfección |
| Kit de transcripción inversa rápida | para la síntesis de ADNc |
| Kit de amplificación de PCR | 2 × Súper SYBR Verde Mezcla maestra qPCR |
2.4 Con respecto a las cuestiones a las que se debe prestar atención en los pasos experimentales específicos:
①proceso de transfección de siRNA
1. Para el cultivo en placas, puede elegir una placa de 24 pocillos, una placa de 12 pocillos o una placa de 6 pocillos (la concentración de ARN media propuesta en cada pocillo de una placa de 24 pocillos es de aproximadamente 100-300 ng/uL), y la densidad de transfección óptima de las células es de hasta un 60 %-80 % más o menos
2. Los pasos de transfección y los requisitos específicos se ajustan estrictamente a las instrucciones.
3. Después de la transfección, las muestras se pueden recolectar dentro de las 24 a 72 horas para la detección de ARNm (RT-PCR) o la detección de proteínas dentro de las 48 a 96 horas (WB)
② Proceso de extracción de ARN
1. Prevenir la contaminación por enzimas exógenas.Incluye principalmente el uso estricto de mascarillas y guantes;usando puntas de pipeta esterilizadas y tubos EP;el agua utilizada en el experimento debe estar libre de ARNasa.
2. Se recomienda hacer el doble de lo sugerido en el kit de extracción rápida, lo que realmente mejorará la pureza y el rendimiento.
3. El líquido de desecho no debe tocar la columna de ARN.
③ Cuantificación de ARN
Después de extraer el ARN, se puede cuantificar directamente con Nanodrop y la lectura mínima puede ser tan baja como 10 ng/ul.
④Proceso de transcripción inversa
1. Debido a la alta sensibilidad de RT-qPCR, se deben hacer al menos 3 pocillos paralelos para cada muestra para evitar que el Ct subsiguiente sea demasiado diferente o que la SD sea demasiado grande para el análisis estadístico.
2. No congele y descongele la mezcla maestra repetidamente.
3. ¡Cada tubo/agujero debe reemplazarse con una punta nueva!¡No utilice continuamente la misma punta de pipeta para añadir muestras!
4. La película adherida a la placa de 96 pocillos después de agregar la muestra debe alisarse con una placa.Lo mejor es centrifugar antes de ponerlo en la máquina, para que el líquido en la pared del tubo pueda fluir hacia abajo y eliminar las burbujas de aire.
⑤Análisis de curva común
| Sin período de crecimiento logarítmico | Posiblemente alta concentración de plantilla |
| Sin valor CT | Pasos incorrectos para detectar señales fluorescentes; degradación de cebadores o sondas: su integridad puede detectarse mediante electroforesis PAGE; cantidad insuficiente de plantilla; degradación de las plantillas: evitar la introducción de impurezas y la congelación y descongelación repetidas en la preparación de muestras; |
| CT>38 | Baja eficiencia de amplificación;El producto de PCR es demasiado largo;varios componentes de la reacción se degradan |
| Curva de amplificación lineal | Las sondas pueden degradarse parcialmente por ciclos repetidos de congelación y descongelación o exposición prolongada a la luz |
| La diferencia en agujeros duplicados es particularmente grande | La solución de reacción no está completamente fundida o la solución de reacción no está mezclada;el baño termal del instrumento PCR está contaminado con sustancias fluorescentes |
2.5 Acerca del análisis de datos
El análisis de datos de qPCR se puede dividir en cuantificación relativa y cuantificación absoluta.Por ejemplo, las células del grupo de tratamiento en comparación con las células del grupo de control,
Cuántas veces cambia el ARNm del gen X, esta es una cuantificación relativa;en un cierto número de células, el ARNm del gen X
Cuántas copias hay, esto es cuantificación absoluta.Normalmente lo que más utilizamos en el laboratorio es el método cuantitativo relativo.Generalmente,el método 2-ΔΔctse usa más en experimentos, por lo que solo este método se presentará en detalle aquí.
Método 2-ΔΔct: El resultado obtenido es la diferencia en la expresión del gen diana en el grupo experimental con respecto al gen diana en el grupo control.Se requiere que las eficiencias de amplificación tanto del gen objetivo como del gen de referencia interno sean cercanas al 100 %, y la desviación relativa no debe exceder el 5 %.
El método de cálculo es el siguiente:
Δct grupo control = valor ct del gen diana en el grupo control – valor ct del gen de referencia interno en el grupo control
Δct grupo experimental = valor ct del gen diana en el grupo experimental – valor ct del gen de referencia interno en el grupo experimental
ΔΔct=Δct grupo experimental-Δct grupo control
Finalmente, calcule el múltiplo de la diferencia en el nivel de expresión:
Change Fold=2-ΔΔct (correspondiente a la función de Excel es POTENCIA)
Productos relacionados:
Hora de publicación: 20-may-2023