Ⅰ. Aumentar la sensibilidad del sistema de reacción:
1. Separe el ARN de alta calidad:
La síntesis exitosa de ADNc proviene de ARN de alta calidad.El ARN de alta calidad debe garantizar al menos un total más largo y no contiene inhibidores que no contengan enzimas de registro, como EDTA o SDS.La calidad del ARN determina el valor máximo de la información de la secuencia que puede transcribir al ADNc.El método general de purificación de ARN es un método escalonado de uso de isocianato/acidofenol.Para evitar la contaminación de la RNasa, el ARN separado de una muestra rica en RNasa (como el páncreas) requiere el almacenamiento de formaldehído para guardar el ARN de alta calidad, lo que es aún más importante para el almacenamiento a largo plazo.El ARN extraído del hígado de rata básicamente se degradó después de una semana de almacenamiento en agua, mientras que el ARN extraído del bazo de rata se mantuvo estable después de tres años de almacenamiento en agua.Además, las transcripciones de más de 4 kb son más sensibles a la degradación de trazas de ARNasa que las transcripciones pequeñas.Para aumentar la estabilidad de la muestra de ARN almacenada, el ARN se puede disolver en una metalamina de iones y se almacena a -70 °C.Thylide utilizado para guardar el ARN no debe contener un objeto misceláneo que degrade el ARN.El ARN, que se deriva del páncreas, se puede guardar en metalamina durante al menos un año.Cuando esté listo para usar el ARN, puede usar los siguientes métodos para precipitar el ARN: agregue NaCl a 0,2 m y 4 veces el volumen de etanol, colóquelo a temperatura ambiente durante 3 a 5 minutos y centrífugo a 10 000 × g durante 5 minutos.
2. Usar transcriptasa inversa sin actividad RNaseH (RNaseH-):
Los inhibidores de la ARNasa a menudo se agregan a las reacciones de transcripción inversa para aumentar la duración y el rendimiento de la síntesis de ADNc.El inhibidor de ARNasa se agrega en la primera reacción de síntesis en cadena en presencia de tampones y agentes reductores como DTT porque el proceso de síntesis de ADNc previo desnaturaliza el inhibidor, liberando así las ARNasas unidas que degradan el ARN.El inhibidor de proteína RNasa solo previene la degradación del ARN por la RNasa A, B, C, y no previene las RNasas en la piel, por lo que se debe tener cuidado de no introducir RNasas de los dedos a pesar del uso de estos inhibidores.
La transcriptasa inversa cataliza la conversión de ARN en ADNc.Tanto M-MLV como AMV tienen actividad RNaseH endógena además de su propia actividad polimerasa.La actividad de la RNasaH compite con la actividad de la polimerasa por cadenas heterocigóticas formadas entre moldes de ARN y cebadores de ADN o cadenas de extensión de ADNc, y degrada cadenas de ARN:ARN en complejos de ADN.Las plantillas de ARN degradadas por la actividad de RNasaH ya no se pueden usar como sustratos efectivos para la síntesis de ADNc, lo que reduce el rendimiento y la duración de la síntesis de ADNc.Por lo tanto, eliminar o reducir en gran medida la actividad de RNasaH de la transcriptasa inversa sería de gran beneficio.
La transcriptasa inversa SuperScriptⅡ, la transcriptasa inversa MMLV de RNaseH- y la transcriptasa inversa thermoScript, AMV de RNaseH- produjeron más ADNc de longitud completa que MMLV y AMV.La sensibilidad de la RT-PCR se ve afectada por la cantidad de ADNc sintetizado.ThermoScript es mucho más sensible que AMV.El tamaño de los productos de RT-PCR está limitado por la capacidad de la transcriptasa inversa para sintetizar ADNc, especialmente cuando se clonan ADNc más grandes.En comparación con MMLV, SuperScripⅡ aumentó significativamente el rendimiento de los productos de RT-PCR largos.La transcriptasa inversa de RNaseH- también aumenta la estabilidad térmica, por lo que la reacción se puede llevar a cabo a temperaturas superiores a las normales de 37-42 ℃.En las condiciones de síntesis sugeridas, se usaron cebadores oligo(dT) y 10 μCi de [alfa-p]dCTP.La producción total de la primera cadena se calculó mediante el método de precipitación TCA.El ADNc de longitud completa se analizó mediante la eliminación de tiras clasificadas por tamaño y el recuento en un gel de agarosa alcalino.
3. Aumente la temperatura de conservación del calor de la transcripción inversa:
Una temperatura de mantenimiento más alta ayuda a abrir la estructura secundaria del ARN y aumenta el rendimiento de la reacción.Para la mayoría de las plantillas de ARN, mantener el ARN y el cebador a 65 °C sin tampón ni sal y luego enfriarlos rápidamente en hielo elimina la mayoría de las estructuras secundarias y permite que los cebadores se unan.Sin embargo, algunas plantillas todavía tienen una estructura secundaria, incluso después de la desnaturalización térmica.La amplificación de estas plantillas difíciles se puede realizar utilizando la transcriptasa inversa ThermoScript y colocando la reacción de la transcriptasa inversa a temperaturas más altas para mejorar la amplificación.Las temperaturas de mantenimiento más altas también pueden aumentar la especificidad, especialmente cuando la síntesis de ADNc se realiza utilizando cebadores específicos de genes (GSPS) (consulte el Capítulo 3).Si usa GSP, asegúrese de que el valor de Tm del cebador sea el mismo que la temperatura de mantenimiento esperada.No utilice cebadores oligo(dT) y aleatorios por encima de 60 ℃.Los cebadores aleatorios deben mantenerse a 25 ℃ durante 10 minutos antes de aumentar a 60 ℃.Además de usar temperaturas de transcripción inversa más altas, la especificidad se puede mejorar transfiriendo directamente la mezcla de cebador/ARN de la temperatura de desnaturalización de 65 ℃ a la temperatura de mantenimiento de la transcripción inversa y agregando una mezcla de reacción 2x precalentada (síntesis de iniciación térmica de ADNc).Este enfoque ayuda a prevenir el apareamiento de bases intermoleculares que se produce a temperaturas más bajas.El uso de un instrumento de PCR simplifica los numerosos cambios de temperatura necesarios para la RT-PCR.
La polimerasa Tth termoestabilizada actúa como ADN polimerasa en presencia de Mg2+ y ARN polimerasa en presencia de Mn2+.Puede mantener el calor hasta 65 ℃.Sin embargo, la presencia de Mn2+ durante la PCR reduce la fidelidad, lo que hace que la polimerasa Tth sea menos adecuada para la amplificación de alta precisión, como la clonación de ADNc.Además, Tth es menos eficiente en la transcripción inversa, lo que reduce la sensibilidad, y dado que una sola enzima puede realizar la transcripción inversa y la PCR, las reacciones de control sin transcripción inversa no se pueden usar para distinguir los productos amplificados de ADNc de los de ADN genómico contaminado.
4. Aditivo que promueve la transcripción inversa:
La adición de aditivos, incluidos glicerina y DMSO, a la primera reacción de síntesis en cadena puede reducir la estabilidad de la doble hebra del ácido nucleico y desenrollar la estructura secundaria del ARN.Se puede agregar hasta un 20 % de glicerina o un 10 % de DMSO sin afectar la actividad de SuperScriptⅡ o MMLV.AMV también puede tolerar hasta un 20% de glicerol sin reducir la actividad.Para maximizar la sensibilidad de la RT-PCR en la reacción de transcripción inversa SuperScriptⅡ, se puede agregar un 10 % de glicerol y aislarlo a 45 ℃.Si se añade 1/10 del producto de la reacción de retrotranscripción a la PCR, la concentración de glicerol en la reacción de amplificación es del 0,4 %, que no es suficiente para inhibir la PCR.
5. Procesamiento de RNaseH:
La sensibilidad se puede mejorar tratando las reacciones de síntesis de cDNA con RNaseH antes de la PCR.Para algunas plantillas, se cree que el ARN en la reacción de síntesis de ADNc evita la unión de los productos amplificados, en cuyo caso el tratamiento con RNaseH puede aumentar la sensibilidad.En general, el tratamiento con RNaseH es necesario para la amplificación de una plantilla diana de cDNA de longitud completa relativamente larga, como la escherosis tuberosa Ⅱ con copia baja.Para esta plantilla difícil, RNaseH mejoró la señal generada por el cDNA sintetizado por SuperScriptⅡ o AMV.Para la mayoría de las reacciones de RT-PCR, el tratamiento con RNaseH es opcional porque el paso de desnaturalización de PCR aislado a 95 ℃ generalmente hidroliza el ARN del complejo ARN: ADN.
6. Métodos mejorados para detectar pequeñas cantidades de ARN:
La RT-PCR es particularmente desafiante cuando solo se dispone de pequeñas cantidades de ARN.La adición de glucógeno como transportador durante la separación del ARN ayuda a aumentar el rendimiento de las muestras pequeñas.Se puede añadir un glucógeno libre de ARNasa al mismo tiempo que Trizol.El glucógeno es soluble en agua y puede permanecer en la fase acuosa con el ARN para ayudar en la precipitación posterior.La concentración recomendada de glucógeno libre de ARNasa es de 250 μg/ml para muestras de menos de 50 mg de tejido o 106 células cultivadas.
La adición de BSA acetilada a las reacciones de transcripción inversa mediante SuperScriptⅡ puede aumentar la sensibilidad y, para pequeñas cantidades de ARN, reducir la cantidad de SuperScriptⅡ y agregar 40 unidades del inhibidor de nucleasa RnaseOut puede mejorar el nivel de detección.Si se utiliza glucógeno en la separación de ARN, aún se recomienda la adición de inhibidores de BSA o RNasa a las reacciones de transcripción inversa mediante SuperScriptⅡ.
Ⅱ. Aumentar la especificidad de la RT-PCR
1. Síntesis de cNDA:
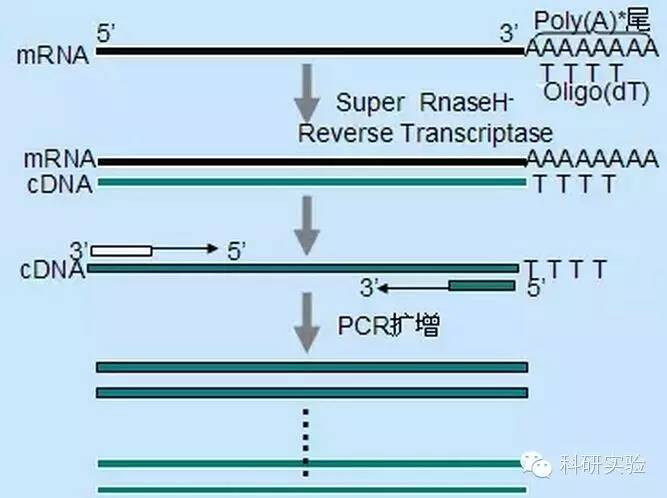
Se pueden usar tres métodos diferentes para iniciar la síntesis de ADNc de la primera cadena, y la especificidad relativa de cada método afecta la cantidad y el tipo de ADNc sintetizado.
El método del cebador aleatorio es el menos específico de los tres métodos.Los cebadores se hibridan en múltiples sitios a lo largo de la transcripción para producir ADNc corto de longitud parcial.Este método se usa a menudo para obtener secuencias terminales 5' y ADNc a partir de plantillas de ARN con regiones estructurales secundarias o con sitios de terminación que la transcriptasa inversa no puede replicar.Para obtener el cDNA más largo, la proporción de cebadores a RNA en cada muestra de RNA debe determinarse empíricamente.La concentración inicial de cebadores aleatorios oscila entre 50 y 250 ng por 20 μl de sistema de reacción.Debido a que el ADNc sintetizado a partir del ARN total usando cebadores aleatorios es principalmente ARN ribosómico, generalmente se selecciona poli(A)+ARN como molde.
La iniciación de oligo(dT) es más específica que los cebadores aleatorios.Se hibrida con la cola poli(A) que se encuentra en el extremo 3′ del ARNm en la mayoría de las células eucariotas.Debido a que el ARN poli(A)+ es aproximadamente del 1 % al 2 % del ARN total, la cantidad y la complejidad del ADNc son mucho menores que si se usaran cebadores aleatorios.Debido a su alta especificidad, el oligo(dT) generalmente no requiere optimización para la proporción de ARN a cebador y selección de poli(A)+.Se recomienda utilizar 0,5 μg de oligo(dT) por 20 μl de sistema de reacción.oligo(dT)12-18 es adecuado para la mayoría de las RT-PCR.El sistema ThermoScript RT-PCR proporciona oligo(dT)20 debido a su buena estabilidad térmica y es adecuado para temperaturas de mantenimiento más altas.
Los cebadores específicos de genes (GSP) son los mejores cebadores específicos para el paso de transcripción inversa.GSP es un oligonucleósido antisentido que puede hibridarse específicamente con secuencias de destino de ARN, en lugar de hibridar todos los ARN como cebadores aleatorios u oligo(dT).Las reglas utilizadas para diseñar cebadores de PCR también se aplican al diseño de la reacción de transcripción inversa GSP.GSP puede tener la misma secuencia que el cebador de amplificación hibridado al final de mRNA3', o GSP puede diseñarse para hibridarse cadena abajo con el cebador de amplificación inversa.Para algunos objetos amplificados, es necesario diseñar más de un cebador antisentido para una RT-PCR exitosa porque la estructura secundaria del ARN objetivo puede evitar que el cebador se una.Se sugiere utilizar 1 pmol de GSP antisentido en el primer sistema de reacción de síntesis en cadena de 20 μl.
2. Aumente la temperatura de conservación del calor de la transcripción inversa:
Para aprovechar al máximo la especificidad de GSP, debe usarse transcriptasa inversa con alta estabilidad térmica.La transcriptasa inversa estable al calor se puede aislar a temperaturas más altas para aumentar el rigor de la reacción.Por ejemplo, si un GSP se recoce a 55 °C, entonces la especificidad del GSP no se utiliza por completo si la transcripción inversa se realiza a 37 °C con poco rigor usando AMV o M-MLV.Sin embargo, SuperScripⅡ y ThermoScript pueden reaccionar a 50 ℃ o más, lo que elimina los productos no específicos producidos a temperaturas más bajas.Para obtener la máxima especificidad, la mezcla de cebador/ARN se puede transferir directamente desde la temperatura de desnaturalización de 65 ℃ a la temperatura de mantenimiento de la transcripción inversa con la adición de una mezcla de reacción 2x precalentada (iniciación térmica de la síntesis de ADNc).Esto ayuda a prevenir el emparejamiento de bases entre moléculas a bajas temperaturas.El uso de un instrumento de PCR simplifica las numerosas transiciones de temperatura necesarias para la RT-PCR.
3. Reducir la contaminación del ADN genómico:
Una dificultad potencial con RT-PCR es que el ARN contamina el ADN genómico.El uso de mejores métodos de separación de ARN, como Trizol Reagent, reduce la contaminación del ADN genómico en las preparaciones de ARN.Para evitar productos producidos a partir de ADN genómico, el ARN se puede tratar con ADNsⅠ de grado de amplificación para eliminar el ADN contaminado antes de la transcripción inversa.Las muestras se mantuvieron a 65 ℃ en EDTA 2,0 mM durante 10 min para terminar la digestión con DNaseⅠ.El EDTA quela los iones de magnesio para evitar la hidrólisis del ARN dependiente de los iones de magnesio que se produce a altas temperaturas.
Para separar el ADNc amplificado del producto de amplificación del ADN genómico, se pueden diseñar cebadores que hibridan por separado con el exón separado.Los productos de PCR derivados del ADNc serán más cortos que los derivados del ADN genómico contaminado.También se realiza un experimento controlado sin transcripción inversa en cada plantilla de ARN para determinar si un fragmento dado es de ADN genómico o de ADNc.Los productos de PCR obtenidos en ausencia de transcripción inversa se derivan del genoma.
Producto relacionado
-El kit de un solo paso permite realizar la transcripción inversa y la PCR en el mismo tubo.Solo necesita agregar ARN de plantilla, cebadores de PCR específicos y ddH sin ARNasa2O.
-El análisis cuantitativo en tiempo real del ARN se puede realizar de forma rápida y precisa.
-El kit utiliza un reactivo de transcripción inversa único de Foregene y la polimerasa de ADN Foregene HotStar Taq combinadas con un sistema de reacción único para mejorar de manera efectiva la eficiencia de amplificación y la especificidad de la reacción.
-El sistema de reacción optimizado hace que la reacción tenga una mayor sensibilidad de detección, una mayor estabilidad térmica y una mejor tolerancia.
-Capacidad eficiente para eliminar gDNA, que puede eliminar gDNA en la plantilla en 2 minutos.
-Sistema de transcripción inversa eficiente, solo toma 15 minutos completar la síntesis de la primera cadena de ADNc.
-Plantillas complejas: las plantillas con alto contenido de GC y estructura secundaria compleja también se pueden revertir con alta eficiencia.
-Sistema de transcripción inversa de alta sensibilidad, las plantillas de nivel pg también pueden obtener ADNc de alta calidad.
-El sistema de transcripción inversa tiene una alta estabilidad térmica, la temperatura de reacción óptima es de 42 ℃ y todavía tiene un buen rendimiento de transcripción inversa a 50 ℃.
Hora de publicación: 07-mar-2023