一、Aumentar la sensibilidad del sistema de reacción:
1. Aislar ARN de alta calidad:
La síntesis exitosa de ADNc proviene de ARN de alta calidad.El ARN de alta calidad debe ser al menos de longitud completa y estar libre de inhibidores de la transcriptasa inversa como EDTA o SDS.La calidad del ARN determina la cantidad máxima de información de secuencia que puede transcribir en ADNc.Un método común de purificación de ARN es un método de un solo paso que utiliza isotiocianato de guanidina/fenol ácido.Para evitar la contaminación por trazas de ARNasa, el ARN aislado de muestras ricas en ARNasa (como el páncreas) debe almacenarse en formaldehído para preservar el ARN de alta calidad, especialmente para el almacenamiento a largo plazo.El ARN extraído del hígado de rata básicamente se degradó después de almacenarse en agua durante una semana, mientras que el ARN extraído del bazo de rata se mantuvo estable después de almacenarse en agua durante 3 años.Además, las transcripciones de más de 4 kb son más sensibles a la degradación por trazas de RNasas que las transcripciones pequeñas.Para aumentar la estabilidad de las muestras de ARN almacenadas, el ARN puede disolverse en formamida desionizada y almacenarse a -70 °C.La formamida utilizada para conservar el ARN debe estar libre de residuos que degradan el ARN.El ARN del páncreas se puede conservar en formamida durante al menos un año.Cuando se prepare para usar ARN, puede usar el siguiente método para precipitar el ARN: agregue NaCl a 0,2 M y 4 veces el volumen de etanol, colóquelo a temperatura ambiente durante 3-5 minutos y centrifugue a 10 000 × g durante 5 minutos.
2. Utilice la transcriptasa inversa RNaseH-inactiva (RNaseH-):
Los inhibidores de la ARNasa a menudo se agregan a las reacciones de transcripción inversa para aumentar la duración y el rendimiento de la síntesis de ADNc.Los inhibidores de RNasa deben agregarse durante la reacción de síntesis de la primera cadena en presencia de un tampón y un agente reductor (como DTT), porque el proceso previo a la síntesis de cDNA desnaturaliza el inhibidor, liberando así la RNasa unida que puede degradar el RNA.Los inhibidores de la proteína RNasa solo previenen la degradación del ARN por la RNasa A, B, C y no previenen la RNasa en la piel, así que tenga cuidado de no introducir la RNasa de sus dedos a pesar del uso de estos inhibidores.
La transcriptasa inversa cataliza la conversión de ARN a ADNc.Tanto M-MLV como AMV tienen actividad RNaseH endógena además de su propia actividad polimerasa.La actividad de la RNasaH y la actividad de la polimerasa compiten entre sí por la cadena híbrida formada entre el molde de ARN y el cebador de ADN o la cadena de extensión de ADNc, y degradan la cadena de ARN en el complejo ARN:ADN.La plantilla de ARN degradada por la actividad de RNasaH ya no puede servir como sustrato eficaz para la síntesis de ADNc, lo que reduce el rendimiento y la duración de la síntesis de ADNc.Por lo tanto, sería beneficioso eliminar o reducir en gran medida la actividad RNasaH de la transcriptasa inversa.。
La transcriptasa inversa SuperScript Ⅱ, RNaseH- MMLV transcriptasa inversa y la transcriptasa inversa thermoScript, RNaseH-AMV, pueden obtener más cantidad y más ADNc de longitud completa que MMLV y AMV.La sensibilidad de la RT-PCR se verá afectada por la cantidad de síntesis de ADNc.ThermoScript es mucho más sensible que AMV.El tamaño de los productos de RT-PCR está limitado por la capacidad de la transcriptasa inversa para sintetizar ADNc, especialmente cuando se clonan ADNc más grandes.En comparación con MMLV, SuperScripⅡ aumentó significativamente el rendimiento de los productos de RT-PCR largos.La transcriptasa inversa RNaseH también tiene una mayor termoestabilidad, por lo que la reacción se puede realizar a temperaturas superiores a los 37-42 °C normales.En las condiciones de síntesis sugeridas, utilice cebador oligo(dT) y 10 μCi de [α-P]dCTP.El rendimiento total de la primera hebra se calculó utilizando el método de precipitación TCA.El ADNc de longitud completa se analizó utilizando bandas clasificadas por tamaño, escindidas y contadas en un gel de agarosa alcalino.
3. Aumente la temperatura de incubación para la transcripción inversa:
Una temperatura de incubación más alta ayuda a abrir la estructura secundaria del ARN, aumentando el rendimiento de la reacción.Para la mayoría de las plantillas de ARN, incubar el ARN y los cebadores a 65 °C sin tampón ni sal, seguido de un enfriamiento rápido en hielo, eliminará la mayoría de las estructuras secundarias y permitirá que los cebadores se unan.Sin embargo, algunas plantillas todavía tienen estructuras secundarias, incluso después de la desnaturalización por calor.La amplificación de estas plantillas difíciles se puede realizar utilizando ThermoScript Reverse Transcriptase y colocando la reacción de transcripción inversa a una temperatura más alta para mejorar la amplificación.Las temperaturas de incubación más altas también pueden aumentar la especificidad, especialmente cuando se utilizan cebadores específicos de genes (GSP) para la síntesis de ADNc (consulte el Capítulo 3).Si usa GSP, asegúrese de que la Tm de los cebadores sea la misma que la temperatura de incubación esperada.No utilice cebadores oligo(dT) y aleatorios por encima de 60 °C.Los cebadores aleatorios requieren incubación a 25 °C durante 10 minutos antes de aumentar a 60 °C.Además de utilizar una temperatura de transcripción inversa más alta, la especificidad también se puede mejorar transfiriendo directamente la mezcla de cebador/ARN de la temperatura de desnaturalización de 65 °C a la temperatura de incubación de transcripción inversa y agregando una mezcla de reacción 2x precalentada (síntesis de inicio en caliente de ADNc) .Este enfoque ayuda a prevenir el apareamiento de bases intermoleculares que se produce a temperaturas más bajas.El cambio de temperatura múltiple requerido para RT-PCR se puede simplificar mediante el uso de un termociclador.
La polimerasa termoestable Tth actúa como ADN polimerasa en presencia de Mg2+ y como ARN polimerasa en presencia de Mn2+.Se puede mantener caliente a una temperatura máxima de 65°C.Sin embargo, la presencia de Mn2+ durante la PCR reduce la fidelidad, lo que hace que la polimerasa Tth sea menos adecuada para la amplificación de alta precisión, como la clonación de ADNc.Además, Tth tiene una eficiencia de transcripción inversa baja, lo que reduce la sensibilidad y, dado que la transcripción inversa y la PCR se pueden realizar con una sola enzima, las reacciones de control sin transcripción inversa no se pueden usar para comparar productos de amplificación de ADNc con ADN genómico contaminante.Los productos de amplificación se separaron.
4. Aditivos que promueven la transcripción inversa:
Se agregan aditivos que incluyen glicerol y DMSO a la reacción de síntesis de la primera hebra, lo que puede reducir la estabilidad de la doble hebra del ácido nucleico y desvincular la estructura secundaria del ARN.Se puede agregar hasta un 20 % de glicerol o un 10 % de DMSO sin afectar la actividad de SuperScript II o MMLV.AMV también puede tolerar hasta un 20% de glicerol sin pérdida de actividad.Para maximizar la sensibilidad de la RT-PCR en la reacción de transcripción inversa SuperScriptⅡ, se puede agregar un 10 % de glicerol e incubar a 45 °C.Si se añade 1/10 del producto de la reacción de transcripción inversa a la PCR, la concentración de glicerol en la reacción de amplificación es del 0,4 %, lo que no es suficiente para inhibir la PCR.
5. Tratamiento de RNaseH:
El tratamiento de las reacciones de síntesis de cDNA con RNaseH antes de la PCR puede aumentar la sensibilidad.Para algunas plantillas, se cree que el ARN en la reacción de síntesis de ADNc impide la unión de los productos de amplificación, en cuyo caso el tratamiento con ARNasaH puede aumentar la sensibilidad.En general, el tratamiento con RNaseH es necesario cuando se amplifican plantillas diana de cDNA de longitud completa más largas, como la escherosis tuberosa II de bajo número de copias.Para esta plantilla difícil, el tratamiento con RNaseH mejoró la señal producida por SuperScript II o ADNc sintetizado por AMV.Para la mayoría de las reacciones de RT-PCR, el tratamiento con RNasaH es opcional, porque el paso de desnaturalización de la PCR a 95 °C generalmente hidroliza el ARN en el complejo ARN:ADN.
6. Mejora del método de detección de ARN pequeño:
La RT-PCR es especialmente desafiante cuando solo se dispone de pequeñas cantidades de ARN.El glucógeno agregado como transportador durante el aislamiento del ARN ayuda a aumentar el rendimiento de las muestras pequeñas.Se puede agregar glucógeno libre de ARNasa al mismo tiempo que se agrega Trizol.El glucógeno es soluble en agua y se puede mantener en la fase acuosa con ARN para ayudar a la precipitación posterior.Para muestras de menos de 50 mg de tejido o 106 células cultivadas, la concentración recomendada de glucógeno libre de ARNasa es de 250 μg/ml.
Agregar BSA acetilado a la reacción de transcripción inversa usando SuperScript II puede aumentar la sensibilidad y, para pequeñas cantidades de ARN, reducir la cantidad de SuperScript II y agregar 40 unidades del inhibidor de nucleasa RNaseOut puede aumentar el nivel de detección.Si se usa glucógeno en el proceso de aislamiento de ARN, aún se recomienda agregar BSA o un inhibidor de RNasa cuando se usa SuperScript II para la reacción de transcripción inversa.
二、Aumentar la especificidad de RT-PCR
1. Síntesis de CND:
La síntesis de ADNc de primera cadena puede iniciarse usando tres métodos diferentes, cuya especificidad relativa afecta la cantidad y el tipo de ADNc sintetizado.
El método del cebador aleatorio fue el menos específico de los tres métodos.Los cebadores se hibridan en múltiples sitios a lo largo de la transcripción, generando ADNc cortos de longitud parcial.Este método se usa con frecuencia para obtener secuencias del extremo 5' y para obtener ADNc a partir de plantillas de ARN con regiones de estructura secundaria o con sitios de terminación que no pueden ser replicados por la transcriptasa inversa.Para obtener el cDNA más largo, la proporción de cebadores a RNA en cada muestra de RNA debe determinarse empíricamente.La concentración inicial de cebadores aleatorios osciló entre 50 y 250 ng por 20 μl de reacción.Dado que el ADNc sintetizado a partir del ARN total utilizando cebadores aleatorios es principalmente ARN ribosómico, generalmente se elige poli(A)+ARN como molde.
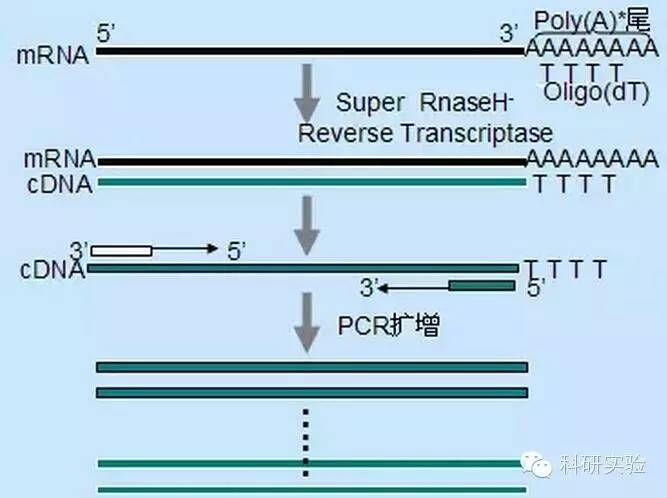
Los cebadores oligo(dT) son más específicos que los cebadores aleatorios.Se hibrida con la cola poli(A) que se encuentra en el extremo 3′ de la mayoría de los mRNA eucarióticos.Debido a que el ARN poli(A)+ es aproximadamente del 1 % al 2 % del ARN total, la cantidad y complejidad del ADNc es mucho menor que con los cebadores aleatorios.Debido a su alta especificidad, oligo(dT) generalmente no requiere optimización de la proporción de ARN a cebadores y selección de poli(A)+.Se recomienda utilizar 0,5 μg de oligo(dT) por 20 μl de sistema de reacción.oligo(dT)12-18 es adecuado para la mayoría de las RT-PCR.El sistema ThermoScript RT-PCR ofrece oligo(dT)20 debido a su mejor estabilidad térmica para temperaturas de incubación más altas.
Los cebadores específicos de genes (GSP) son los cebadores más específicos para el paso de transcripción inversa.GSP es un oligonucleótido antisentido que se puede hibridar específicamente con la secuencia diana de ARN, a diferencia de los cebadores aleatorios u oligo(dT), que se hibridan con todos los ARN.Las mismas reglas utilizadas para diseñar cebadores de PCR se aplican al diseño de GSP en reacciones de transcripción inversa.El GSP puede tener la misma secuencia que el cebador de amplificación que se hibrida con el extremo 3' del ARNm, o el GSP puede diseñarse para que se hibrida cadena abajo del cebador de amplificación inversa.Para algunos sujetos amplificados, es necesario diseñar más de un cebador antisentido para que la RT-PCR tenga éxito porque la estructura secundaria del ARN objetivo puede impedir la unión del cebador.Se recomienda utilizar 1 pmol de GSP antisentido en una reacción de síntesis de la primera cadena de 20 μl.
2. Aumente la temperatura de incubación para la transcripción inversa:
Para aprovechar al máximo la ventaja total de la especificidad de GSP, debe usarse una transcriptasa inversa con mayor termoestabilidad.Las transcriptasas inversas termoestables se pueden incubar a temperaturas más altas para aumentar la rigurosidad de la reacción.Por ejemplo, si un GSP se hibrida a 55 °C, la especificidad del GSP no se utilizará por completo si se usa AMV o M-MLV para la transcripción inversa a una rigurosidad baja de 37 °C.Sin embargo, SuperScript II y ThermoScript pueden reaccionar a 50 °C o más, lo que eliminará los productos no específicos generados a temperaturas más bajas.Para obtener la máxima especificidad, la mezcla de cebador/ARN puede transferirse directamente desde la temperatura de desnaturalización de 65 °C a la temperatura de incubación de transcripción inversa y agregarse a una mezcla de reacción 2x precalentada (inicio en caliente de la síntesis de ADNc).Esto ayuda a prevenir el emparejamiento de bases intermoleculares a bajas temperaturas.Las múltiples transiciones de temperatura necesarias para la RT-PCR se pueden simplificar mediante el uso de un termociclador.
3. Reduce la contaminación del ADN genómico:
Una posible dificultad que se encuentra con la RT-PCR es la contaminación del ADN genómico en el ARN.El uso de un buen método de aislamiento de ARN, como Trizol Reagent, reducirá la cantidad de ADN genómico que contamina la preparación de ARN.Para evitar productos derivados del ADN genómico, el ARN se puede tratar con ADNasa I de grado de amplificación para eliminar el ADN contaminante antes de la transcripción inversa.La digestión con ADNasa I se terminó incubando las muestras en EDTA 2,0 mM durante 10 minutos a 65°C.EDTA puede quelar iones de magnesio, evitando la hidrólisis de ARN dependiente de iones de magnesio a altas temperaturas.
Con el fin de separar el ADNc amplificado de los productos de amplificación de ADN genómico contaminantes, se pueden diseñar cebadores para que cada uno se hibrida con exones separados.Los productos de PCR derivados del ADNc serán más cortos que los derivados del ADN genómico contaminado.Además, se realizó un experimento de control sin transcripción inversa en cada plantilla de ARN para determinar si un fragmento determinado se derivaba de ADN genómico o ADNc.El producto de PCR obtenido sin transcripción inversa se deriva del genoma.
Hora de publicación: 16-may-2023








