PCR, múltiple PCR, PCR in situ, PCR inversa, RT-PCR, qPCR(1)–PCR
Clasificaremos los conceptos, pasos y detalles de varios PCR
Ⅰ. PCR
La reacción en cadena de la polimerasa, conocida como PCR, es una tecnología de biología molecular que se utiliza para agrandar fragmentos de ADN específicos.Puede considerarse como una replicación especial del ADN in vitro.La ADN polimerasa (ADN polimerasa I) se descubrió ya en 1955, y el fragmento Klenow de E. Coli, que tiene valor experimental y practicidad, fue descubierto por el Dr. H. Klenow a principios de la década de 1970, pero debido a que esta enzima no tolera la temperatura, la alta temperatura puede degenerarla, por lo que no cumple con la reacción en cadena de la polimerasa con la degeneración a alta temperatura.Las enzimas que se usan hoy en día (llamadas Taq polimerasa), fueron aisladas de Thermus aquaticus, una bacteria de aguas termales en 1976. Su característica es que puede resistir altas temperaturas y es una enzima ideal, pero se usa ampliamente después de la década de 1980.El concepto original del prototipo primitivo original de PCR es similar a la reparación y copia de genes, propuesta por el Dr. KJell Kleppe en 1971. Publicó la primera copia de genes simple y de corto plazo (similar a las dos primeras reacciones de ciclo de PCR).El PCR desarrollado hoy fue desarrollado por el Dr. Kary B. Mullis en 1983. El Dr. Mullis prestó servicios a empresas de PE ese año, por lo que PE tiene un estatus especial en la industria de PCR.El Dr. Mullis publicó oficialmente el primer artículo relacionado con Saiki y otros en 1985. Desde entonces, el uso de PCR es de miles de millas por día, y se puede decir que la calidad de los artículos relacionados hace que muchos otros métodos de investigación sean desagradables.Posteriormente, la tecnología PCR se utiliza ampliamente en la investigación científica biológica y aplicaciones clínicas, convirtiéndose en la tecnología más importante de la investigación en biología molecular.Mullis también ganó el Premio Nobel de Química en 1993.
PCRPrincipio
El principio básico de la tecnología PCR es similar al proceso de replicación natural del ADN, y su especificidad depende del cebador del oligonucleótido que es complementario a ambos extremos de la secuencia objetivo.La PCR se compone de tres pasos básicos de reacción de degeneración-recocido-extensión: ①Degeneración del ADN molde: después de que el ADN molde se calienta a unos 93 °C durante un cierto período de tiempo, la solución de ADN dual para el ADN de doble cadena formado por la amplificación por PCR del ADN molde que se va, lo convierte en una sola cadena para que pueda combinarse con el cebador para preparar la siguiente ronda de reacción.②El recocido (compuesto) de la plantilla de ADN y el cebador: después de que la plantilla de ADN se calienta y se degenera en una sola cadena, la temperatura desciende a unos 55 °C.La secuencia complementaria del cebador y la plantilla de ADN monocatenario.③La extensión del cebador: plantilla de ADN: la unión del cebador se basa en la acción de la polimerasa TaqDNA, con dNTP como materia prima de reacción.Mantenga el principio de replicación, sintetice una nueva cadena de copia semi-reservada que complemente la cadena de ADN plantilla, y repita el ciclo degeneración-recocido-extensión tres procesos pueden obtener más "cadena de copia semi-reservada", y esta nueva cadena está disponible nuevamente. Conviértase en una plantilla para el próximo ciclo.Se necesitan de 2 a 4 minutos para completar el ciclo, el gen objetivo se puede amplificar varios millones de veces en 2 a 3 horas.
EstándarPCRSistema de reacción
| ADN polimerasa Taq | 2,5 l |
| Mg2+ | 1.5mmol/L |
| Tampón de amplificación 10× | 10μl |
| 4 mezclas de dNTP | 200μl |
| Plantilla de ADN | 0.1~2μg |
| Cebador | 10~100μl |
| Agregue agua hirviendo doble o triple | 100 l |
Cinco elementos de la reacción de PCR
Hay principalmente cinco tipos de sustancias involucradas en la reacción de PCR, a saber, cebador, enzima, dNTP, plantilla y tampón (se requiere Mg2+).[Procedimiento PCR]
El proceso de PCR estándar se divide en tres pasos
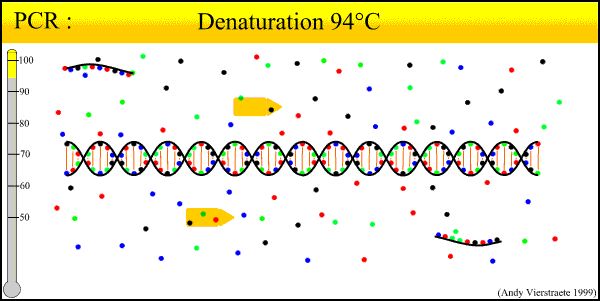
1. Degeneración del ADN (90°C-96°C): Plantillas de ADN de doble cadena bajo acción térmica, los enlaces de hidrógeno se rompen y forman un ADN de cadena única.
2. Recocido (25 ℃ -65 ℃): la temperatura del sistema se reduce, el cebador se combina con la plantilla de ADN para formar una doble cadena local.
3. Extensión (70 ℃ -75 ℃): Bajo la acción de la enzima Taq (alrededor de 72 °C, la mejor actividad), se usa dNTP como materia prima, se extiende desde el extremo 5 'del cebador → extremo 3', la síntesis y la plantilla se complementan entre sí cadena de ADN.
Cada ciclo se desnaturaliza, recoce y extiende, duplicando el contenido de ADN.En la actualidad, debido al área de amplificación corta, algunas PCR se pueden replicar en muy poco tiempo incluso si la actividad de la enzima Taq no es óptima, por lo que se puede cambiar a dos pasos, es decir, la hibridación y la extensión se pueden realizar a 60 °C-65 °C al mismo tiempo.Con el fin de reducir el proceso de elevación y enfriamiento y mejorar la velocidad de respuesta.
Características de la reacción de PCR
● alta especificidad
Los factores decisivos específicos de la respuesta de la PCR son: ①La combinación específica del cebador y la plantilla de ADN.②El principio del emparejamiento de bases.③La lealtad de la reacción de síntesis de la polimerasa TaqDNA.④La especificidad y el carácter conservador del gen diana.
La combinación correcta de cebadores y plantillas es la clave.La unión del cebador y la plantilla y la extensión de la cadena del cebador se basan en el principio de coincidencia de bases alcalinas.La lealtad de las reacciones de síntesis de polimerasa y la resistencia a altas temperaturas de la ADN polimerasa Taq para hacer que la unión (compuesto) de la plantilla y el cebador en la reacción se pueda realizar a una temperatura más alta.La especificidad de la combinación aumenta considerablemente.El clip puede mantener un alto grado de corrección.Al seleccionar una región genética diana con un alto nivel de conservación y un alto nivel de conservación, su especificidad es mayor.
● Alta sensibilidad
El volumen de producción de productos de PCR aumenta por índice, que puede expandir la plantilla inicial de Picker (PG = 10-12) para aumentar el nivel del microcontrolador al nivel de microgramos (μg = -6).Se pueden detectar células diana a partir de 1 millón de células;en la detección de virus, la sensibilidad de la PCR puede llegar a 3 RFU (unidades formadas por puntos vacíos);la tasa mínima de detección en la ciencia bacteriana es de 3 bacterias.
● Sencillo y Rápido
La reflexión de PCR utiliza una polimerasa de ADN Taq de alta temperatura, que agrega la solución de reacción de una sola vez, es decir, una reacción de degeneración-recocido-extensión en la solución de amplificación de ADN y el recipiente del baño de agua.Generalmente, la reacción de amplificación se completa en 2 a 4 horas.Los productos aumentados generalmente se analizan con una espada eléctrica y no tienen que usar isótopos, no hay contaminación radiactiva y son de fácil promoción.
● La pureza de la muestra es baja
No hay necesidad de separar virus o bacterias y células de cultivo.Los productos brutos de ADN y el ARN se pueden utilizar como amplificadores.La detección de amplificación de ADN se puede usar directamente usando muestras clínicas como sangre, líquido corporal, líquido para lavar la tos, cabello, células y tejido vivo.
PCRproblemas comunes
● Falso negativo, sin bandas amplificadas
Las etapas clave de la reacción de PCR incluyen: ① preparación de ácidos nucleicos molde, ② calidad y especificidad de los cebadores, ③ calidad de las enzimas ④ condiciones del ciclo de PCR.Encontrar la razón también debe analizarse y estudiarse para los enlaces anteriores.
Plantillas: ① La plantilla contiene varias proteínas, ② La plantilla contiene un inhibidor de la enzima Taq, ③ La proteína de la plantilla no se elimina, especialmente el grupo de proteínas en el cromosoma.⑤ La degeneración del ácido nucleico del desminador no es exhaustiva.Cuando la calidad de las enzimas y los cebadores es buena, no hay banda de amplificación, que es probablemente el tratamiento digestivo de los especímenes.Hay algo mal con el proceso de extracción de ácido nucleico de plantilla, por lo que para preparar una solución de digestión eficaz y estable, su procedimiento debe corregirse y no cambiarse arbitrariamente.
Inactivación de enzimas: debe usarse una nueva enzima o ambas enzimas, la antigua y la nueva, para analizar si la actividad enzimática se pierde o es insuficiente, lo que da lugar a falsos negativos.Cabe señalar que a veces se olvida la enzima Taq o el bromuro de etidio.
Cebador: la calidad del cebador, la concentración del cebador y si la concentración de los dos cebadores es simétrica.Es una razón común para la falla de la PCR o el aumento de la banda no es ideal y tiende a difundirse.Hay problemas con la calidad de los cebadores de algunos números de lote.Los dos cebadores tienen una concentración alta y una concentración baja, lo que provoca una amplificación asimétrica de baja eficiencia.Las contramedidas son: ① Seleccionar un buen cebador para sintetizar unidades.② La concentración de la imprimación no solo depende del valor de OD, sino que también presta atención al líquido original de la imprimación para hacer electroforesis en gel de azúcar de agar.Debe haber una zona de tira de imprimación, y el brillo de las dos imprimaciones debe ser generalmente consistente.Belt, la PCR puede fallar en este momento y debe resolverse con la unidad de síntesis de cebadores.Si una imprimación es alta, el brillo es bajo y su concentración debe equilibrarse cuando se diluye.③ La imprimación debe pagarse y almacenarse en una concentración alta para evitar la congelación múltiple o la refrigeración a largo plazo de partes del refrigerador, lo que hará que la imprimación se deteriore y se degrade.④ El diseño del cebador no es razonable, como que la longitud del cebador es insuficiente y se forma el grupo di entre los cebadores.
Concentración de Mg2+: La concentración de iones de Mg2+ tiene un gran impacto en la eficiencia de la amplificación por PCR.Una concentración excesiva puede reducir el sexo opuesto de la amplificación por PCR.Si la concentración es demasiado baja, la salida de amplificación de PCR incluso hará que la amplificación de PCR falle sin la banda de expansión.
Cambio de volumen de reacción: el volumen utilizado en la amplificación por PCR es de 20 ul, 30 ul y 50 ul o 100 ul, el gran volumen de la solicitud de amplificación por PCR se establece de acuerdo con diferentes propósitos de investigación científica y pruebas clínicas.Después de hacer pequeños volúmenes como 20ul, es necesario hacer una condición de cordón al hacer el tamaño, de lo contrario fallará.
Razones físicas: la transformación es muy importante para la amplificación por PCR.Si la temperatura de degeneración es baja, el tiempo de degeneración es corto, es probable que se produzcan falsos negativos;una temperatura de recocido demasiado baja puede causar una amplificación no específica y reducir la eficiencia de la amplificación específica.Afecta en gran medida a la combinación de cebadores y plantillas para reducir la eficiencia de la amplificación por PCR.En ocasiones es necesario utilizar termómetros estándar para detectar la variabilidad, el recocido y la temperatura extendida en el cocedor de extensión o hidrosoluble, lo cual es una de las razones del fracaso de la PCR.
Variantes de la secuencia diana: Si se produce la secuencia diana, una mutación o deleción, la combinación del prototipo y el molde se combina, o por falta de una secuencia diana, el cebador y el molde perderán la secuencia complementaria, y su amplificación por PCR no será exitosa.
● Falso positivo
La banda de amplificación de PCR parece consistente con la banda de la secuencia objetivo y, a veces, su banda es más nítida y más alta.
El diseño del cebador no es apropiado: la secuencia de amplificación seleccionada y la secuencia de amplificación sin propósito tienen homólogos, por lo que cuando se realiza la amplificación por PCR, los productos de PCR amplificados son secuencias sin propósito.La secuencia diana es demasiado corta o el cebador es demasiado corto y es propenso a falsos positivos.Necesita ser rediseñado.
Contaminación cruzada de la secuencia diana o de los productos de amplificación: hay dos motivos para esta contaminación: en primer lugar, la contaminación cruzada del genoma completo o de grandes segmentos, lo que da lugar a falsos positivos.Este tipo de falso positivo se puede resolver mediante los siguientes métodos: Sea cuidadoso y cuidadoso durante la operación para evitar que la secuencia objetivo se inhale en la pistola de muestra o salpique fuera del tubo centrífugo.A excepción de las enzimas y las sustancias que no pueden soportar altas temperaturas, todos los reactivos o equipos deben desinfectarse con alta presión.Las tuberías centrífugas y las muestras deben usarse al mismo tiempo.Cuando sea necesario, antes de agregar las muestras, el tubo de reacción y el reactivo se exponen a los rayos ultravioleta para destruir el ácido nucleico existente.Segundo, pequeños fragmentos en la contaminación del aire.Estos pequeños fragmentos son más cortos que la secuencia diana, pero tienen cierta homología.Se puede empalmar entre sí.Después de complementar los cebadores, el producto de PCR se puede expandir, lo que provocará la producción de falsos positivos.Se puede utilizar para reducir o eliminar el método de PCR de nido.
● Aparece una banda de amplificación no específica
Las bandas que aparecieron después de la amplificación por PCR son inconsistentes con el tamaño esperado, o son grandes o pequeñas, o al mismo tiempo, o al mismo tiempo, bandas de amplificación específicas y bandas de amplificación no específicas.La aparición de bandas no específicas es: Primero, los cebadores son complementarios incompletos a la secuencia objetivo, o la polimerización del cebador para formar un grupo di.La segunda es que la concentración de iones MG2+ es demasiado alta, la temperatura de hibridación es demasiado baja y el número de ciclos de PCR está relacionado.En segundo lugar, la calidad y cantidad de enzimas.A menudo, las enzimas de algunas fuentes son propensas a bandas no especiales y no se producen las enzimas de la otra fuente.A veces también se produce una amplificación no específica de las enzimas.Las contramedidas son: atractivos rediseñados si es necesario.Reduzca la cantidad de enzima o reemplace la enzima de otra fuente.Reduzca la cantidad de primaria, aumente la cantidad de plantillas adecuadamente y reduzca la cantidad de ciclos.Aumente adecuadamente la temperatura de recocido o use el método de dos puntos de temperatura (degeneración a 93 °C, recocido y extensión a unos 65 °C).
● Apariencia de estopa escamosa o cinta manchada
La amplificación por PCR a veces parece estar aplicada o descascarada o como un cinturón tipo alfombra.Por la razón, debido a la cantidad excesiva de enzimas o la mala calidad de la enzima, la concentración de dNTP es demasiado alta, la concentración de Mg2+ es demasiado alta, la temperatura de recocido es demasiado baja y la cantidad de ciclos es demasiado.Las contramedidas son: ① Reducir la cantidad de enzimas o cambiar la enzima de otra fuente.②Reducir la concentración de dNTP ③Reducir adecuadamente la concentración de Mg2+.④Aumente la cantidad de plantillas y reduzca el número de ciclos.
Productos relacionados
◮ Mayor fidelidad: 6 veces más que la enzima Taq ordinaria;
◮ Mayor velocidad de amplificación
◮ Más adaptabilidad de plantilla
◮ Mayor eficiencia de amplificación
◮ La tolerancia ambiental es más fuerte: se coloca a 37°C durante una semana, manteniendo más del 90% de actividad;
◮ Tiene actividad ADN polimerasa 5'→3' y actividad exonucleasa 5'→3', sin actividad exonucleasa 3'→5'.
El sistema de reacción único y la ADN polimerasa Taq de alta eficiencia hacen que la reacción de PCR tenga una mayor eficiencia de amplificación, especificidad y sensibilidad.
RT-qPCR Easyᵀᴹ (Un paso)-SYBR Green I
◮ El kit de un solo paso hace que la transcripción inversa y la qPCR sean dos reacciones en el mismo tubo, solo es necesario agregar ARN de plantilla, cebadores de PCR específicos y ddH sin ARNasa2O.
◮ El kit puede analizar cuantitativamente de manera rápida y eficiente el ARN viral o rastrear el ARN.
◮ El kit utiliza un reactivo de transcripción inversa único de Foregene y la polimerasa de ADN Foregene HotStar Taq combinadas con un sistema de reacción único para mejorar de manera efectiva la eficiencia de amplificación y la especificidad de la reacción.
◮ El sistema de reacción optimizado hace que la reacción tenga una mayor sensibilidad de detección, una mayor estabilidad térmica y una mejor tolerancia.
◮ RT-qPCR FácilTM(Un paso): el kit SYBR Green I viene con un colorante de referencia interno ROX, que se puede usar para eliminar el fondo de la señal y los errores de señal entre pozos, lo cual es conveniente para que los clientes lo usen en diferentes modelos de instrumentos PCR cuantitativos.
RT fácilTMII (Premezcla maestra para Síntesis de ADNc de primera cadena paraPCR en tiempo real)
-Capacidad eficiente para eliminar gDNA, que puede eliminar gDNA en la plantilla en 2 minutos.
-Sistema de transcripción inversa eficiente, solo toma 15 minutos completar la síntesis de la primera cadena de ADNc.
-Plantillas complejas: las plantillas con alto contenido de GC y estructura secundaria compleja también se pueden revertir con alta eficiencia.
-Sistema de transcripción inversa de alta sensibilidad, las plantillas de nivel pg también pueden obtener ADNc de alta calidad.
-El sistema de transcripción inversa tiene una alta estabilidad térmica, la temperatura de reacción óptima es de 42 ℃ y todavía tiene un buen rendimiento de transcripción inversa a 50 ℃.
Hora de publicación: 18-mar-2023