La vacuna de ARNm de Pfizer para la COVID ha reavivado la pasión por utilizar el ácido ribonucleico (ARN) como diana terapéutica.Sin embargo, apuntar al ARN con moléculas pequeñas es extremadamente desafiante.
El ARN solo tiene cuatro componentes básicos: adenina (A), citosina (C), guanina (G) y uracilo (U) que reemplaza a la timina (T) que se encuentra en el ADN.Esto hace que la selectividad de las drogas sea un obstáculo casi insuperable.Por el contrario, hay 22 aminoácidos naturales que componen las proteínas, lo que explica por qué la mayoría de los fármacos dirigidos a las proteínas tienen una selectividad relativamente buena.
Estructura y función del ARN
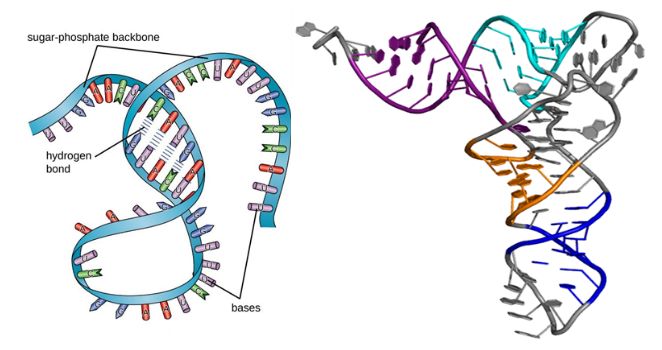
Al igual que las proteínas, las moléculas de ARN tienen estructuras secundarias y terciarias, como se muestra en la siguiente figura.Aunque son macromoléculas de una sola cadena, su estructura secundaria toma forma cuando el emparejamiento de bases provoca protuberancias, bucles y hélices.Luego, el plegamiento tridimensional conduce a la estructura terciaria del ARN, que es esencial para su estabilidad y función.
Figura 1. Estructura del ARN
Hay tres tipos de ARN:
- ARN mensajero (ARNm)transcribe la información genética del ADN y se transfiere como secuencia de bases al ribosoma;yo
- ARN ribosómico (ARNr)es parte de los orgánulos sintetizadores de proteínas llamados ribosomas, que se exportan al citoplasma y ayudan a traducir la información del ARNm en proteínas;
- ARN de transferencia (ARNt)es el enlace entre el ARNm y la cadena de aminoácidos que forma la proteína.
Dirigirse al ARN como diana terapéutica es muy atractivo.Se ha descubierto que solo el 1,5 % de nuestro genoma se traduce finalmente en proteína, mientras que entre el 70 % y el 90 % se transcribe en ARN.Las moléculas de ARN son las más importantes para todos los organismos vivos.Según el “dogma central” de Francis Crick, la función más crítica del ARN es traducir la información genética del ADN en proteínas.Además, las moléculas de ARN también tienen otras funciones, entre ellas:
- Actuando como moléculas adaptadoras en la síntesis de proteínas;yo
- Sirviendo como mensajero entre el ADN y el ribosoma;yo
- Son portadores de información genética en todas las células vivas;yo
- Promover la selección ribosomal de los aminoácidos correctos, que es necesario para sintetizar nuevas proteínasen vivo.
antibióticos
A pesar de haber sido descubierto ya en la década de 1940, el mecanismo de acción de muchos antibióticos no se aclaró hasta finales de la década de 1980.Se ha descubierto que una gran proporción de antibióticos actúan uniéndose a los ribosomas bacterianos para evitar que produzcan las proteínas apropiadas, matando así a las bacterias.
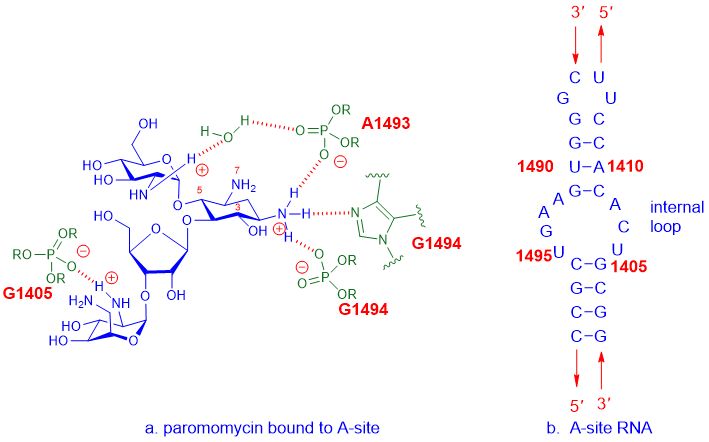
Por ejemplo, los antibióticos aminoglucósidos se unen al sitio A del ARNr 16S, que es parte de la subunidad del ribosoma 30S, y luego interfieren con la síntesis de proteínas para interferir con el crecimiento bacteriano, lo que finalmente conduce a la muerte celular.El sitio A se refiere al sitio aminoacyl, también conocido como el sitio aceptor de tRNA.La interacción detallada entre los fármacos aminoglucósidos, comoparomomicina, y el sitio A deE. coliEl ARN se muestra a continuación.
Figura 2. La interacción entre la paromomicina y el sitio A deE. coliARN
Desafortunadamente, muchos inhibidores del sitio A, incluidos los fármacos aminoglucósidos, tienen problemas de seguridad, como nefrotoxicidad, ototoxicidad irreversible específica y dependiente de la dosis.Estas toxicidades son el resultado de la falta de selectividad de los fármacos aminoglucósidos para reconocer moléculas pequeñas de ARN.
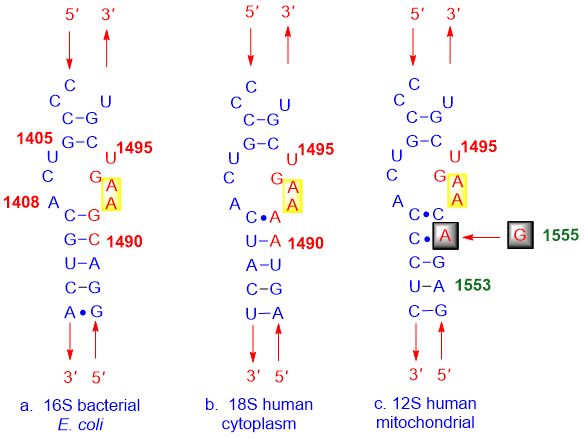
Como se muestra en la siguiente figura: (a) la estructura de la bacteria, (b) la membrana celular humana y (c) el sitio A mitocondrial humano son muy similares, lo que hace que los inhibidores del sitio A se unan a todos ellos.
Figura 3. La unión del inhibidor del sitio A no selectivo
Los antibióticos de tetraciclina también inhiben el sitio A del ARNr.Inhiben selectivamente la síntesis de proteínas bacterianas al unirse de manera reversible a una región helicoidal (H34) en la subunidad 30S complejada con Mg2+.
Por otro lado, los antibióticos macrólidos se unen cerca del sitio de salida (sitio E) del túnel del ribosoma bacteriano para péptidos nacientes (NPET) y lo bloquean parcialmente, inhibiendo así la síntesis de proteínas bacterianas.Finalmente, los antibióticos de oxazolidinona tales comolinezolid(Zyvox) se unen a una hendidura profunda en la subunidad ribosomal 50S bacteriana, que está rodeada por nucleótidos de ARNr 23S.
Oligonucleótidos antisentido (ASO)
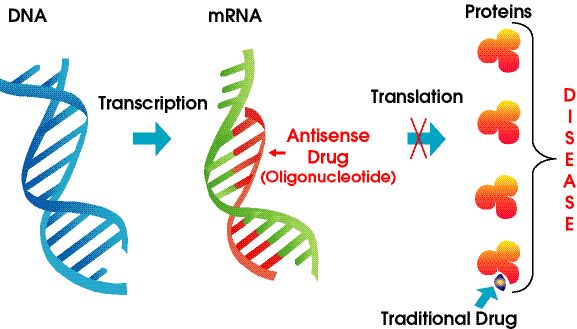
Los fármacos antisentido son polímeros de ácido nucleico modificados químicamente que se dirigen al ARN.Se basan en el emparejamiento de bases de Watson-Crick para unirse al ARNm objetivo, lo que resulta en el silenciamiento génico, el bloqueo estérico o la alteración del corte y empalme.Los ASO pueden interactuar con los pre-ARN en el núcleo celular y los ARNm maduros en el citoplasma.Pueden apuntar a exones, intrones y regiones no traducidas (UTR).Hasta la fecha, la FDA ha aprobado más de una docena de medicamentos ASO.
Figura 4. Tecnología antisentido
Fármacos de molécula pequeña dirigidos al ARN
En 2015, Novartis informó que había descubierto un regulador de empalme SMN2 llamado Branaplam, que mejora la asociación de U1-pre-mRNA y rescata ratones SMA.
Por otro lado, Risdiplam (Evrysdi) de PTC/Roche fue aprobado por la FDA en 2020 para el tratamiento de la AME.Al igual que Branaplam, Risdiplam también actúa regulando el empalme de genes SMN2 relevantes para producir proteínas SMN funcionales.
degradadores de ARN
RBM significa proteína con motivo de unión a ARN.Esencialmente, la indol sulfonamida es un adhesivo molecular.Recluta selectivamente RBM39 a la ubiquitina ligasa CRL4-DCAF15 E3, promoviendo la poliubiquitinación de RBM39 y la degradación de proteínas.El agotamiento genético o la degradación mediada por sulfonamidas de RBM39 induce anomalías significativas en el empalme de todo el genoma, lo que en última instancia conduce a la muerte celular.
Los RNA-PROTAC se desarrollan para degradar las proteínas de unión a RNA (RBP).PROTAC utiliza un conector para conectar el ligando de ligasa E3 al ligando de ARN, que se une al ARN y las prácticas comerciales restrictivas.Dado que RBP contiene dominios estructurales que pueden unirse a secuencias de oligonucleótidos específicas, RNA-PROTAC utiliza una secuencia de oligonucleótidos como ligando para la proteína de interés (POI).El resultado final es la degradación de las prácticas comerciales restrictivas.
Recientemente, el profesor Matthew Disney de la Institución Scripps de Oceanografía inventó el ARNQuimeras dirigidas a ribonucleasas (RiboTAC).RiboTAC es una molécula heterofuncional que conecta un ligando de ARNasa L y un ligando de ARN con un enlazador.Puede reclutar específicamente RNasa L endógena para objetivos de ARN específicos y luego eliminar con éxito el ARN utilizando el mecanismo de descomposición del ácido nucleico celular (RNasa L).
A medida que los investigadores aprendan más sobre la interacción entre las moléculas pequeñas y los objetivos de ARN, en el futuro surgirán más medicamentos que utilicen este método.
Hora de publicación: 02-ago-2023