Material de partida: ARN
La PCR de transcripción inversa cuantitativa (RT-qPCR) es un método experimental utilizado en experimentos de PCR que utilizan ARN como material de partida.En este método, el ARN total o ARN mensajero (ARNm) se transcribe primero en ADN complementario (ADNc) mediante transcriptasa inversa.Posteriormente, se realizó una reacción de qPCR usando el ADNc como molde.RT-qPCR se ha utilizado en una variedad de aplicaciones de biología molecular, incluido el análisis de expresión génica, la validación de interferencia de ARN, la validación de micromatrices, la detección de patógenos, las pruebas genéticas y la investigación de enfermedades.
Métodos de uno y dos pasos para RT-qPCR
La RT-qPCR se puede lograr mediante un método de uno o dos pasos.La RT-qPCR de un paso combina la transcripción inversa y la amplificación por PCR, lo que permite que la transcriptasa inversa y la ADN polimerasa completen la reacción en el mismo tubo en las mismas condiciones de tampón.La RT-qPCR de un paso solo requiere el uso de cebadores específicos de secuencia.En la RT-qPCR de dos pasos, la transcripción inversa y la amplificación por PCR se realizan en dos tubos, utilizando diferentes tampones optimizados, condiciones de reacción y estrategias de diseño de cebadores.
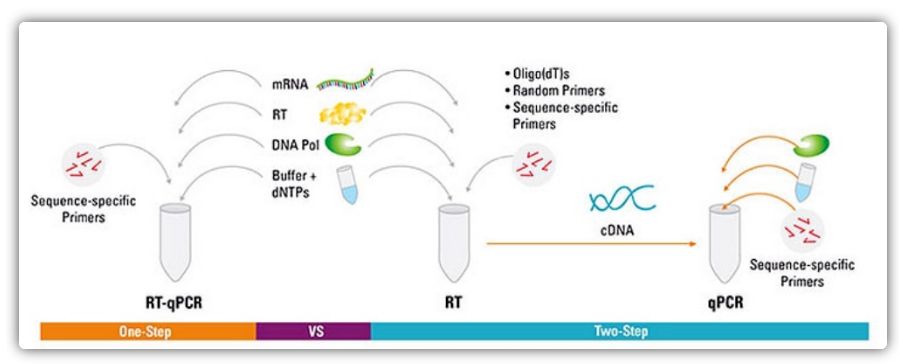
| Ventaja | Desventaja | |
| Un paso | Este método tiene menos error experimental ya que ambas reacciones se realizan en un tubo.
Menos pasos de pipeteo reducen el riesgo de contaminación
Adecuado para amplificación/detección de alto rendimiento, rápido y reproducible | Las reacciones de dos pasos no se pueden optimizar por separado
Dado que las condiciones de reacción se ven comprometidas al combinar la reacción de dos pasos, la sensibilidad no es tan buena como la del método de dos pasos.
El número de objetivos detectados por una sola muestra es pequeño |
| Dos pasos | Capacidad para crear bibliotecas de cDNA estables que pueden almacenarse durante largos períodos de tiempo y usarse en múltiples reacciones
Los genes diana y los genes de referencia se pueden amplificar a partir de la misma biblioteca de ADNc sin necesidad de múltiples bibliotecas de ADNc
Tampones de reacción y condiciones de reacción que permiten la optimización de corridas de reacción individuales
Selección flexible de condiciones de activación | El uso de varios tubos y más pasos de pipeteo aumenta el riesgo de contaminación del ADN, y consume mucho tiempo.
Requiere más optimización que el método de un solo paso |
Productos relacionados:
RT-qPCR Easyᵀᴹ (un paso)-SYBR Green I
RT-qPCR Easyᵀᴹ (un paso)-Taqman
RT Easyᵀᴹ I Master Premix para la síntesis de CDNA de primera cadena
PCR en tiempo real Easyᵀᴹ-SYBR Green I Kit
PCR en tiempo real Easyᵀᴹ-Taqman
Selección de ARN total y ARNm
Al diseñar un experimento de RT-qPCR, es importante decidir si usar ARN total o ARNm purificado como plantilla para la transcripción inversa.Aunque el ARNm puede proporcionar una sensibilidad ligeramente mayor, todavía se usa con frecuencia el ARN total.La razón de esto es que el ARN total tiene una ventaja más importante como material de partida que el ARNm.En primer lugar, el proceso requiere menos pasos de purificación, lo que garantiza una mejor recuperación cuantitativa de la plantilla y una mejor normalización de los resultados con respecto al número de células iniciales.En segundo lugar, evita el paso de enriquecimiento de mRNA, lo que puede evitar la posibilidad de resultados sesgados debido a diferentes recuperaciones de diferentes mRNA.En general, dado que en la mayoría de las aplicaciones la cuantificación relativa del gen diana es más importante que la sensibilidad absoluta de la detección, el ARN total es más adecuado en la mayoría de los casos.
cebador de transcripción inversa
En el método de dos pasos, se pueden usar tres métodos diferentes para cebar la reacción de ADNc: cebadores oligo(dT), cebadores aleatorios o cebadores específicos de secuencia.Normalmente, los cebadores oligo(dT) y los cebadores aleatorios se usan en combinación.Estos cebadores se hibridan con la cadena de ARNm molde y proporcionan a la transcriptasa inversa un punto de partida para la síntesis.
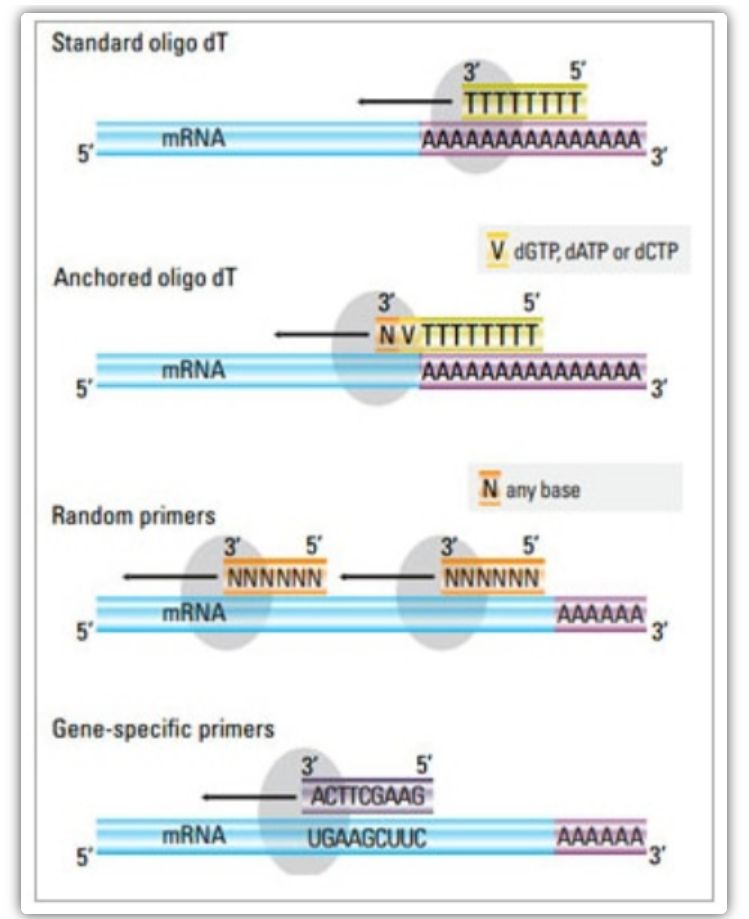
| Selección de imprimación | Estructura y función | Ventaja | Desventaja |
| Cebador oligo(dT) (o cebador oligo(dT) anclado) | Recocido extendido a residuos de timina en la cola poli(A) del ARNm;El cebador oligo(dT) de anclaje contiene una G, C o A en el extremo 3′ (sitio de anclaje) | Síntesis de ADNc de longitud completa a partir de ARNm de cola poli(A)
Aplicable cuando hay menos material de partida disponible
El sitio de anclaje asegura que el cebador oligo(dT) se una a la cola 5′ poli(A) del ARNm | Solo apto para amplificar genes con colas poli(A)
Obtener ADNc truncado del sitio de cebado*2 en poli(A)
Sesgado para unirse al extremo 3′*
*Esta posibilidad se minimiza si se utilizan cebadores oligo(dT) anclados |
| imprimación al azar
| De 6 a 9 bases de longitud, que pueden hibridarse en múltiples sitios durante la transcripción de ARN | Recocido a todos los ARN (tRNA, rRNA y mRNA)
Adecuado para transcripciones con una estructura secundaria significativa o cuando hay menos material de partida disponible
Alto rendimiento de ADNc | El cDNA se transcribe de forma inversa a partir de todo el RNA, lo que generalmente no se desea y puede diluir la señal del mRNA objetivo.
obtener ADNc truncado |
| cebadores específicos de secuencia | Cebadores personalizados dirigidos a secuencias de ARNm específicas | biblioteca específica de ADNc
Mejorar la sensibilidad
Uso de cebadores de qPCR inversa | Solo limitado a la síntesis de un solo gen objetivo. |
La transcriptasa inversa
La transcriptasa inversa es una enzima que utiliza ARN para sintetizar ADN.Algunas transcriptasas inversas tienen actividad RNasa y pueden degradar cadenas de ARN en cadenas híbridas de ARN-ADN después de la transcripción.Si no tiene actividad enzimática de RNase, se puede agregar RNaseH para una mayor eficiencia de qPCR.Las enzimas comúnmente utilizadas incluyen la transcriptasa inversa del virus de la leucemia murina de Moloney y la transcriptasa inversa del virus del mieloblastoma aviar.Para RT-qPCR, es ideal elegir una transcriptasa inversa con mayor termoestabilidad, de modo que la síntesis de cDNA se pueda realizar a temperaturas más altas, lo que garantiza la transcripción exitosa de RNA con una estructura secundaria más alta, mientras mantiene su actividad completa durante la reacción, lo que da como resultado mayores rendimientos de cDNA.
Productos relacionados:
Transcriptasa inversa Foreasy M-MLV
Actividad de ARNasa H de la transcriptasa inversa
RNaseH es capaz de degradar cadenas de ARN de dúplex de ARN-ADN, lo que permite la síntesis eficiente de ADN de doble cadena.Sin embargo, cuando se usa ARNm largo como molde, el ARN puede degradarse prematuramente, lo que da como resultado un ADNc truncado.Por lo tanto, a menudo es beneficioso minimizar la actividad de RNaseH durante la clonación de cDNA si se desea la síntesis de transcritos largos.Por el contrario, las transcriptasas inversas con actividad de RNasa H suelen ser beneficiosas para las aplicaciones de qPCR porque mejoran la fusión de los dúplex de ARN-ADN durante el primer ciclo de PCR.
Primer diseño
Los cebadores de PCR utilizados para el paso de qPCR en RT-qPCR deben diseñarse idealmente para abarcar una unión exón-exón, donde un cebador de amplificación podría potencialmente abarcar un límite exón-intrón real.Dado que las secuencias de ADN genómico que contienen intrones no se amplifican, este diseño reduce el riesgo de amplificación de falsos positivos debido a la contaminación del ADN genómico.
Si los cebadores no pueden diseñarse para separar exones o límites exón-exón, puede ser necesario tratar las muestras de ARN con DNasa I libre de RNasa o dsDNasa para eliminar la contaminación del ADN genómico.
Control RT-qPCR
Debe incluirse un control negativo de transcripción inversa (control -RT) en todos los experimentos de RT-qPCR para detectar contaminación de ADN (como ADN genómico o productos de PCR de reacciones anteriores).Este control contiene todos los componentes de la reacción excepto la transcriptasa inversa.Dado que la transcripción inversa no se produce con este control, si se observa amplificación por PCR, lo más probable es que haya contaminación por ADN.
Hora de publicación: 02-ago-2022








