Después de que el erudito estadounidense Eric S. Lander propusiera formalmente el polimorfismo de nucleótido único (SNP) como marcador molecular de tercera generación en 1996, SNP se ha utilizado ampliamente en el análisis de asociación de rasgos económicos, la construcción de mapas de enlaces genéticos biológicos y la detección de genes patógenos humanos., Diagnóstico y predicción del riesgo de enfermedades, detección individualizada de drogas y otros campos de investigación biológica y médica.En el campo del mejoramiento de cultivos comerciales, la detección de SNP puede realizar una selección temprana de los rasgos requeridos.Esta selección tiene las características de alta precisión y puede evitar efectivamente la interferencia de la morfología y los factores ambientales, lo que acorta en gran medida el proceso de reproducción.Por lo tanto, SNP juega un papel muy importante en el campo de la investigación básica.
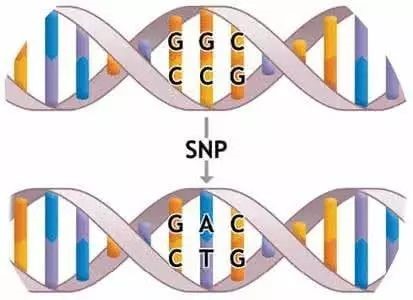
El polimorfismo de un solo nucleótido (Single Nucleotide Polymorphism, SNP) se refiere al fenómeno de que existen diferencias de un solo nucleótido en la misma posición en la secuencia de ADN de individuos de la misma o diferente especie.La inserción, eliminación, conversión e inversión de una sola base pueden causar esta diferencia.En el pasado, la definición de SNP era diferente a la de mutación.Un locus variante requiere que la frecuencia de uno de los alelos en la población sea superior al 1% para ser definido como un locus SNP.Sin embargo, con la expansión de las teorías biológicas modernas y la aplicación de la tecnología, la frecuencia alélica ya no es una condición necesaria para limitar la definición de SNP.Según los datos de variación de un solo nucleótido incluidos en la base de datos de polimorfismos de un solo nucleótido (dbSNP) del Centro Nacional de Información Biotecnológica (NCBI), también se incluyen inserción/eliminación de baja frecuencia, variación de microsatélites, etc.
En el cuerpo humano, la frecuencia de SNP es del 0,1%.En otras palabras, hay un promedio de un sitio SNP por cada 1000 pares de bases.Aunque la frecuencia de ocurrencia es relativamente alta, no todos los sitios SNP pueden ser marcadores candidatos relacionados con los rasgos.Esto está relacionado principalmente con la ubicación donde se produce el SNP.
Teóricamente, SNP puede ocurrir en cualquier parte de la secuencia del genoma.Los SNP que ocurren en la región codificante pueden producir mutaciones sinónimas y mutaciones no sinónimas, es decir, el aminoácido cambia o no cambia antes y después de la mutación.El aminoácido cambiado generalmente hace que la cadena peptídica pierda su función original (mutación sin sentido) y también puede provocar el aborto de la traducción (mutación sin sentido).Los SNP que ocurren en regiones no codificantes y regiones intergénicas pueden afectar el empalme del ARNm, la composición de la secuencia del ARN no codificante y la eficiencia de unión de los factores de transcripción y el ADN.La relación específica se muestra en la figura:
Tipos de SNP:
Varios métodos comunes de tipificación de SNP y su comparación
De acuerdo con diferentes principios, los métodos comunes de detección de SNP se dividen en las siguientes categorías:
Comparación de clasificaciones de métodos de detección
Nota: En la tabla se enumeran los métodos de detección de SNP más comunes que se utilizan actualmente, otros métodos de detección, como hibridación de sitio específico (ASH), extensión de cebador de sitio específico (ASPE), extensión de base única (SBCE), corte de sitio específico (ASC), tecnología de chips genéticos, tecnología de espectrometría de masas, etc. no se han clasificado ni comparado.
El costo y el tiempo de purificación de ácido nucleico en los varios métodos de detección de SNP comunes anteriores son inevitables.Sin embargo, los kits relacionados basados en la tecnología de PCR directa de Foregene pueden realizar directamente la amplificación de PCR o qPCR en muestras no purificadas, lo que brinda una comodidad sin precedentes para la detección de SNP.
Los productos de la serie PCR directa de Foregene omiten de manera simple y aproximada los pasos de purificación de muestras, lo que reduce en gran medida el tiempo y el costo necesarios para preparar plantillas.La polimerasa Taq única tiene una excelente capacidad de amplificación y puede tolerar una variedad de inhibidores de entornos de amplificación complejos.Estas características proporcionan una garantía técnica para la obtención de productos específicos de alto rendimiento. Kits Foregene Direct PCR/qPCR para varios tipos de muestras, tales como: tejidos animales (cola de rata, pez cebra, etc.), hojas de plantas, semillas (incluyendo muestras de polisacáridos y polifenoles), etc.
Hora de publicación: 23-jul-2021