En mi opinión, varias invenciones revolucionarias en la historia de la tecnología de detección son la tecnología de inmunomarcaje basada en el principio de unión específica antígeno-anticuerpo, la tecnología PCR y la tecnología de secuenciación.Hoy hablaremos de la tecnología PCR.De acuerdo con la evolución de la tecnología PCR, las personas habitualmente dividen la tecnología PCR en tres generaciones: tecnología PCR ordinaria, tecnología PCR cuantitativa fluorescente en tiempo real y tecnología PCR digital.
Ctécnica común de PCR

KARY MULLIS (1944.12.28-2019.8.7)
Kary Mullis inventó la reacción en cadena de la polimerasa (polimerasa en cadena, PCR) en 1983. Se dice que cuando conducía a su novia, de repente tuvo un destello de inspiración y pensó en el principio de la PCR (sobre los beneficios de conducir).Kary Mullis recibió el Premio Nobel de Química en 1993. The New York Times comentó: "Muy original y significativo, casi dividiendo la biología en eras previas a la PCR y posteriores a la PCR.
El principio de la PCR: bajo la catálisis de la ADN polimerasa, el ADN de la hebra madre se usa como molde, y el cebador específico se usa como punto de partida de la extensión, y el ADN de la hebra hija complementario al ADN molde de la hebra madre se copia in vitro mediante desnaturalización, hibridación, extensión y otros pasos.Es una tecnología de amplificación de síntesis de ADN in vitro, que puede amplificar rápida y específicamente cualquier ADN objetivo in vitro.
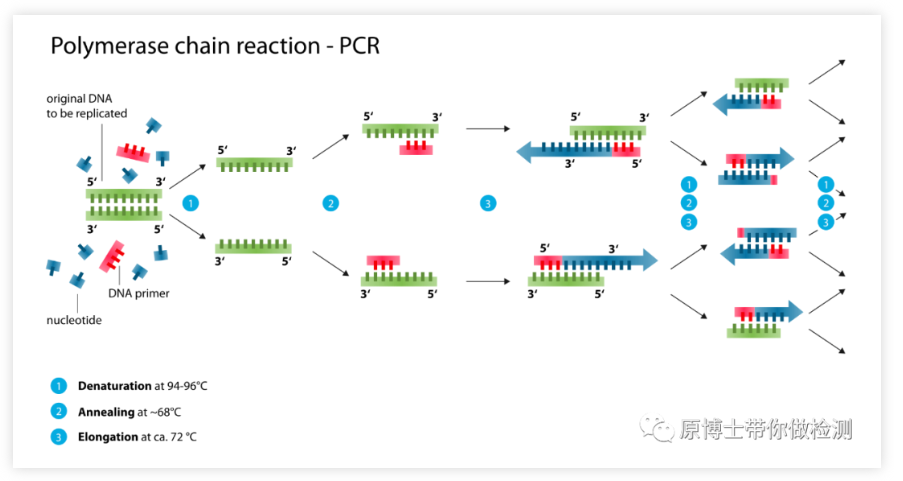
Ventajas de la PCR ordinaria
1.Método clásico, estándares nacionales e internacionales completos
2.Menor costo de los reactivos del instrumento
3.Los productos de PCR se pueden recuperar para otros experimentos de biología molecular
Máquina de PCR Foregene recomendada: https://www.foreivd.com/foreamp-sn-695-series-thermal-cycler-96-wells-pcr-machine-product/
Productos relacionados: https://www.foreivd.com/pcr-herotm-with-dye-product/
Desventajas de la PCR ordinaria
1.fácil de contaminar
2.operación engorrosa
3.solo análisis cualitativo
4.Sensibilidad moderada
5.Hay amplificación no específica, y cuando la banda no específica es del mismo tamaño que la banda objetivo, no se puede distinguir
CPCR basada en electroforesis apilar
En respuesta a las deficiencias de la PCR ordinaria, algunos fabricantes han introducido instrumentos basados en el principio de la electroforesis capilar.El paso de electroforesis después de la amplificación por PCR se completa en el capilar.La sensibilidad es mayor, y se puede distinguir la diferencia de varias bases y MAERKER puede calcular la amplificación.contenido del productoLa desventaja es que el producto de PCR aún debe abrirse y colocarse en el instrumento, y aún existe un gran riesgo de contaminación.

CapilarEelectroforesis
2. Tecnología de PCR cuantitativa fluorescente en tiempo real (PCR cuantitativa en tiempo real, qPCR)La PCR cuantitativa fluorescente, también llamada PCR en tiempo real, es una nueva tecnología cuantitativa de ácido nucleico desarrollada por PE (Perkin Elmer) en 1995. La historia del desarrollo de la PCR cuantitativa fluorescente es una historia de luchas conmovedoras de gigantes como ABI, Roche y Bio-Rad.Si te interesa, puedes echarle un vistazo.Esta técnica es actualmente la técnica de PCR semicuantitativa más madura y más utilizada.
Máquina qPCR recomendada: https://www.foreivd.com/mini-real-time-pcr-system-forequant-sf2sf4-product/
Método de colorante fluorescente (SYBR Green I):SYBR Green I es el colorante de unión al ADN más utilizado para la PCR cuantitativa, que se une de forma no específica al ADN de doble cadena.En estado libre, SYBR Green emite una fluorescencia débil, pero una vez unido al ADN de doble cadena, su fluorescencia aumenta 1000 veces.Por lo tanto, la señal fluorescente total emitida por una reacción es proporcional a la cantidad de ADN de doble cadena presente y aumentará con el aumento del producto amplificado.Dado que el tinte se une de forma no específica al ADN de doble cadena, se pueden generar resultados falsos positivos.
Productos relacionados: https://www.foreivd.com/real-time-pcr-easytm-sybr-green-i-kit-product/
Método de sonda fluorescente (tecnología Taqman): DuranteAmplificación por PCR, se añade una sonda fluorescente específica al mismo tiempo que un par de cebadores.La sonda es un oligonucleótido lineal, con un grupo informador fluorescente y un grupo extintor fluorescente respectivamente marcados en ambos extremos.Cuando la sonda está intacta, la señal fluorescente emitida por el grupo informador es absorbida por el grupo extintor y la detección No hay señal fluorescente;Durante la amplificación por PCR (en la etapa de extensión), la actividad Dicer 5'-3' de la enzima Taq digerirá y degradará la sonda, de modo que el grupo fluorescente reportero y el grupo fluorescente extintor se separen, de modo que el sistema de monitoreo de fluorescencia pueda recibir la señal fluorescente, es decir, cada vez que se amplifica una cadena de ADN, se forma una molécula fluorescente, que realiza la sincronización completa de la acumulación de señales fluorescentes y la formación de productos de PCR.El método de sonda Taqman es el método de detección más utilizado en la detección clínica.
Productos relacionados: https://www.foreivd.com/quickeasy%e1%b5%80%e1%b4%b9-real-time-pcr-kit-taqman-product/
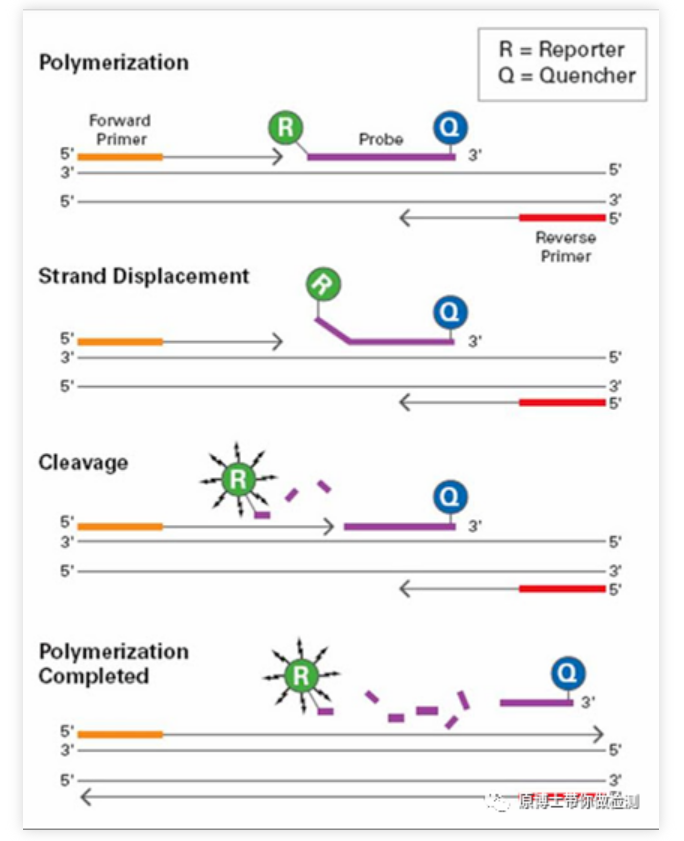
Ventajas de la qPCR
1.El método está maduro y el equipo de apoyo y los reactivos están completos.
2.Coste medio de los reactivos
3.fácil de usar
4.Alta sensibilidad y especificidad de detección
Desventajas de qPCR
La mutación del gen objetivo conduce a una detección perdida.
No se puede determinar el resultado de detección de la plantilla de baja concentración.
Hay un gran error cuando se utiliza la curva estándar para la detección cuantitativa.
3. Tecnología de PCR digital (PCR digital, dPCR)
La PCR digital es una técnica para la cuantificación absoluta de moléculas de ácido nucleico.En comparación con la qPCR, la PCR digital puede leer directamente el número de moléculas de ADN/ARN, que es la cuantificación absoluta de las moléculas de ácido nucleico en la muestra inicial.En 1999, Bert Vogelstein y Kenneth W. Kin-zler propusieron formalmente el concepto de dPCR.
En 2006, Fluidigm fue el primero en producir un instrumento comercial de dPCR basado en un chip.En 2009, Life Technologies lanzó los sistemas OpenArray y QuantStudio 12K Flex dPCR.En 2013, Life Technologies lanzó el sistema QuantStudio 3DdPCR, que utiliza tecnología de chips microfluídicos a nanoescala de alta densidad para distribuir uniformemente las muestras en 20 000 células individuales.en el pozo de reacción.
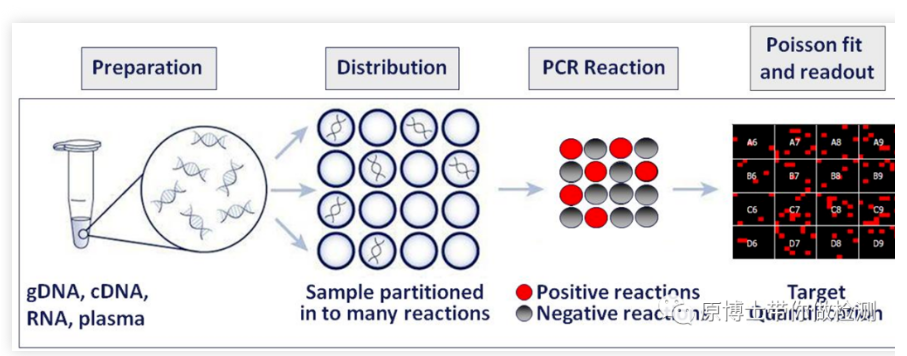
En 2011, Bio-Rad lanzó el instrumento QX100 dPCR basado en gotas, que utiliza tecnología de agua en aceite para distribuir uniformemente la muestra a 20 000 gotas de agua en aceite y utiliza un analizador de gotas para analizar las gotas.En 2012, RainDance lanzó el instrumento RainDrop dPCR, impulsado por gas a alta presión, para dividir cada sistema de reacción estándar en una emulsión de reacción que contiene de 1 millón a 10 millones de microgotas a nivel de picolitros.
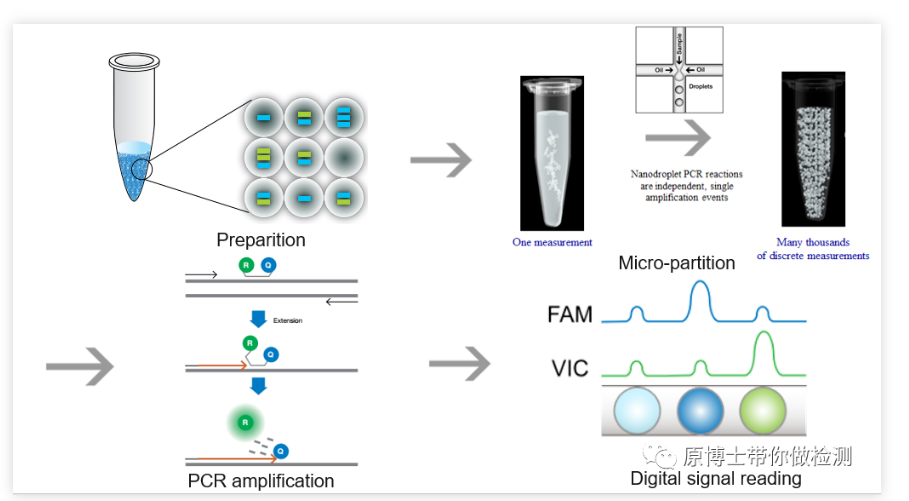
Hasta ahora, la PCR digital ha formado dos facciones principales, tipo chip y tipo gota.No importa qué tipo de PCR digital, sus principios básicos son la dilución limitada, la PCR de punto final y la distribución de Poisson.El sistema de reacción de PCR estándar que contiene plantillas de ácido nucleico se divide uniformemente en decenas de miles de reacciones de PCR, que se distribuyen en chips o microgotas, de modo que cada reacción contenga la mayor cantidad posible de una molécula de plantilla, y se realiza una reacción de PCR de plantilla de molécula única.Mediante la lectura de la fluorescencia se cuenta la presencia o ausencia de la señal, y se realiza la cuantificación absoluta previa calibración de la distribución estadística de Poisson.
Las siguientes son las características de varias plataformas PCR digitales que he utilizado:
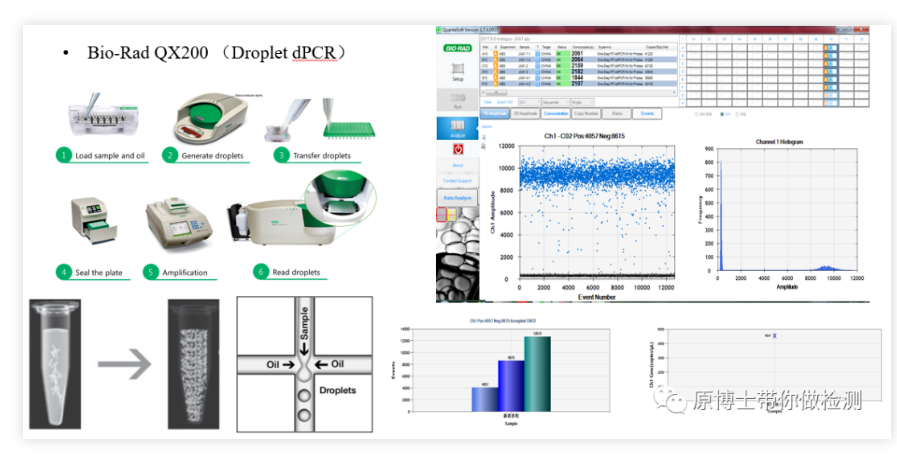
1. PCR digital de gotas Bio-Rad QX200 Bio-RadQX200 es una plataforma de PCR digital muy clásica, el proceso de detección básico: el generador de gotas genera 20,000 muestras. Las microgotas de agua en aceite se amplifican en una máquina de PCR común y, finalmente, la señal de fluorescencia de cada microgota se lee con un lector de microgotas.La operación es más complicada y el riesgo de contaminación es medio.
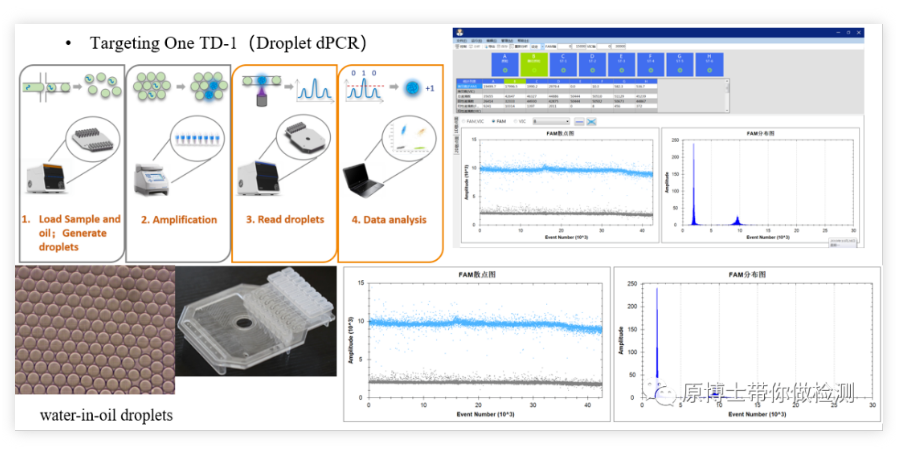
PCR digital de microgotas Xinyi TD1Xinyi TD1 es una plataforma de PCR digital doméstica, el proceso de detección básico: genera 30,000-50,000 gotas de agua en aceite a través de un generador de gotas, amplifica en un instrumento de PCR común y finalmente pasa El lector de gotas lee la señal fluorescente de cada gota.Tanto la generación de gotas como la lectura en esta plataforma se realizan en un chip dedicado con bajo riesgo de contaminación.
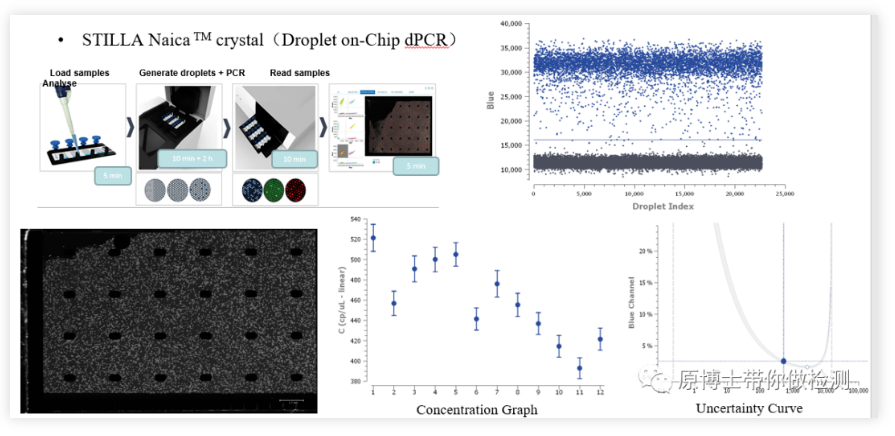
PCR digital con chip de microgotas STILLA NaicaSTILLA Naica es una plataforma PCR digital relativamente nueva.El proceso de detección básico es: agregue la solución de reacción al chip, coloque el chip en el sistema de generación y amplificación de microgotas y genere 30,000 microgotas.Se esparce en el chip y la amplificación por PCR se completa en el chip.Luego, el chip amplificado se transfiere al sistema de análisis de lectura de microgotas y la señal fluorescente se lee tomando fotografías.Dado que todo el proceso tiene lugar en un chip cerrado, el riesgo de contaminación es bajo.
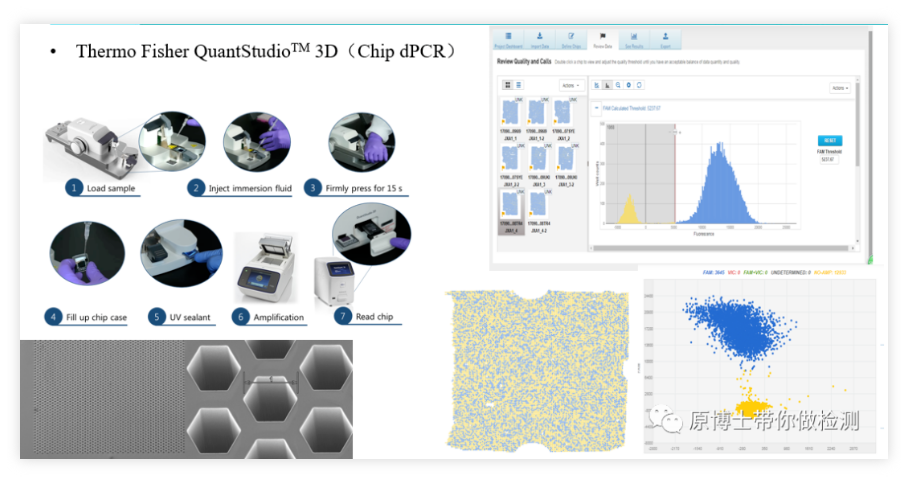
4. PCR digital con chip 3D ThermoFisher QuantStudio
ThermoFisher QuantStudio 3D es otra plataforma clásica de PCR digital basada en chips.Su proceso de detección básico es: agregar la solución de reacción en el esparcidor y esparcir la solución de reacción uniformemente en el chip con 20 000 micropocillos a través del esparcidor., coloque el chip en la máquina PCR para amplificar, y finalmente coloque el chip en el lector y tome una foto para leer la señal fluorescente.La operación es relativamente complicada, y todo el proceso se lleva a cabo en un chip cerrado, y el riesgo de contaminación es bajo.
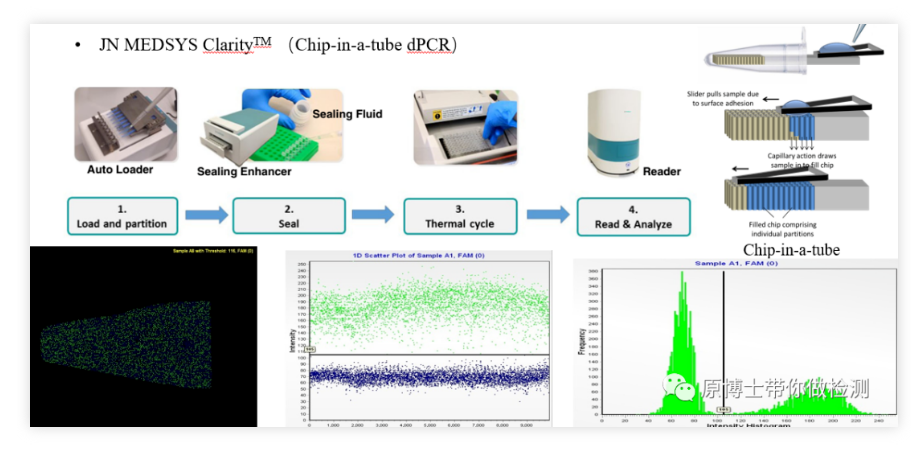
5. PCR digital con chip JN MEDSYS Clarity
JN MEDSYS Clarity es una plataforma de PCR digital tipo chip relativamente nueva.Su proceso de detección básico es: agregue la solución de reacción en el aplicador y distribuya la solución de reacción uniformemente en 10,000 tubos de PCR fijados en el tubo de PCR a través del aplicador.En el chip microporoso, la solución de reacción ingresa al chip a través de la acción capilar, y el tubo de PCR con el chip se coloca en la máquina de PCR para amplificación y, finalmente, el chip se coloca en el lector para leer la señal fluorescente tomando una foto.La operación es más complicada.El riesgo de contaminación es bajo.
Los parámetros de cada plataforma PCR digital se resumen a continuación:
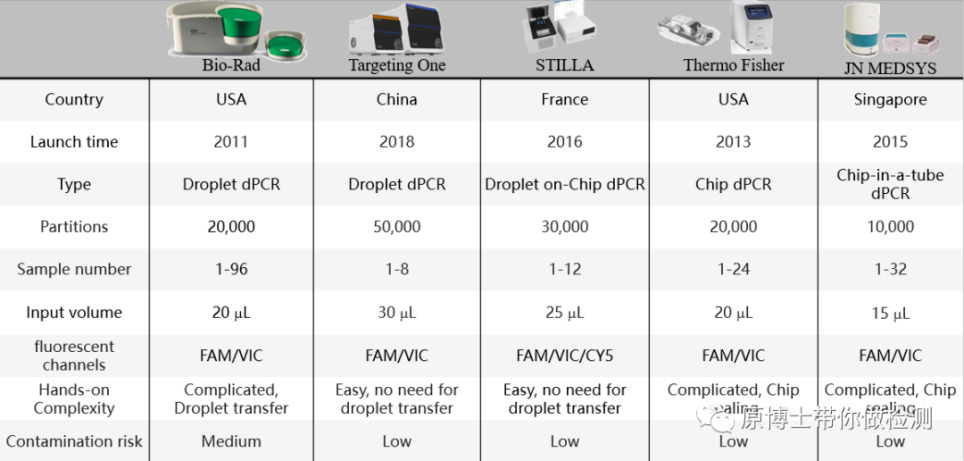
Los indicadores de evaluación de la plataforma PCR digital son: el número de unidades divididas, el número de canales fluorescentes, la complejidad de la operación y el riesgo de contaminación.Pero lo más importante es la precisión de detección.Una forma de evaluar las plataformas de PCR digital es usar múltiples plataformas de PCR digitales para verificarse entre sí, y otra forma es usar sustancias estándar con valores precisos.
Ventajas de la dPCR
1.Lograr la cuantificación absoluta
2.Mayor sensibilidad y especificidad
3.Puede detectar muestras de copia baja
Desventajas de dPCR1. Equipo y reactivos costosos 2. Operación complicada y tiempo de detección largo 3. Rango de detección estrecho
En la actualidad, las tres generaciones de tecnología PCR tienen sus propias ventajas y desventajas, y cada una tiene sus propios campos de aplicación, y no es una relación que una generación reemplace a la otra.El avance continuo de la tecnología ha inyectado nueva vitalidad a la tecnología PCR, permitiéndole desbloquear una dirección de aplicación tras otra, haciendo que la detección de ácido nucleico sea más conveniente y precisa.
Fuente: el Dr. Yuan te lleva a la prueba
Productos Recomendados:
Hora de publicación: 18-nov-2022











